AbstractPURPOSE:The purpose of this study was to examine changes of gene transcripts following 4- and 8-week resistance exercise training in rat skeletal muscle using large-scale BeadArray containing probe sets for 22,523 genes.
METHODS:Forty-eight male Sprague-Dawley rats were enrolled and into control (CON, n=16) and and exercise (resistance training, RT, n=32) groups. After 4- and 8-weeks of resistance exercise, based upon lifting weight of individual rats, RT subsequently was separated into high performance group (H-RT4, n=8; H-RT8, n=8) and low performance group (L-RT4, n=8; L-RT8, n=8). Rats were assigned to RT were trained to climb a 1-m vertical (85 degree incline) ladder with weights secured to their tails, and they performed 1 set of 10 times of 3 days per week for 8 weeks progressively. Skeletal muscle was taken from the flexor halucis longus (FHL) after 4- and 8-weeks exercise. After separating the total RNA of each group, large-scale gene expression was analyzed by BeadArray using Ill-unina RatRef-12 Expression BeadChip, and qPCR was used to inspect BeadArray data. We selected genes given the criteria that detection p-value of <.01, M-value {M=log2(condition)-log2(reference)} to >1.0, and DiffScore to >30.
RESULTS:The expression of 20 genes significantly increased 4 weeks after resistance exercise compared to L-RT4, and the expression of four genes decreased, including genes with unknown functions. At 8 weeks, the expression of one gene significantly increased compared to L-RT8 and that of one decreased. These genes involved in acute phase response (Hp, Ahsg), anti-inflammatory response (Ambp), C21-steroid hormone biosynthesis (Timp1), heme oxidation (Hmox1), muscle development (Myh6), regulation of cell growth (Igfbp1), vitamin D metabolism (Gc), calcium-mediated signaling (Dscr1), gluconeogenesis (Atf3), regulation of transcription (Nr4a3), and learning and memory (Egr1).
CONCLUSIONS:This is the first study to use BeadArray to examine the global mRNA expression in rat skeletal muscle after resistance ladder training. We found expression genes of rat skeletal muscle based on the resistance exercise performance level using BeadArray. These results may help to explain previously reported physiological changes of skeletal muscle and suggest new avenues for further investigation.
서론다형성(polymorphism)과 같은 유전적 변이(genetic variants)는 엘리트 운동선수들의 운동수행능력에 미치는 잠재적인 영향을 이해하기 위한 필수적인 요소로 알려져 왔다. American College of Sports Medicine (ACSM)에서 출판하고 있는 “Medicine & Science in Sports & Exercise”저널에서는 2001년부터 2009년까지 “The Human Gene Map for Performance and Health-Related Fitness Phenotypes”, 2010년부터 현재까지 “Advances in Exercise, Fitness, and Performance Genomics”이란 주제로 운동, 건강, 체력, 수행력 관련 유전자에 대한 연구 결과를 매년 업데이트하여 발표하고 있다. 지금까지 약 200개 이상의 가장 일반적인 DNA 염기서열변이인 유전자 다형성(single nucleotide polymorphism, SNPs)이 운동수행력(performance), 체력(fitness), 특성(traits)과 관련이 있는 것으로 보고되었으며[1], Ahmetov와 Fedotovskaya [2]은 적어도 120개의 유전적 마커(genetic markers)들 중 77개 유전자가 지구성 수행력과 관련이 있고, 43개가 유전자가 근력과 파워와 관련이 있는 것으로 제시하였다. 그러나 이러한 연구결과들은 대부분 다형성과 유전자의 유전형(genotype)을 기초로 단순히 운동수행능력과의 관련성을 조사하였거나 특정 운동수행 후 관련 표현형(phenotype)과 비교한 연구들이거나 특정 유전자에 대한 정량 분석을 통하여 발현 여부를 확인한 연구들이다. 아직까지 microarray 기술과 같은 대규모 유전자 발현양상 분석 툴을 이용한 운동수행능력 관련 후보 유전자군 발굴 연구는 이루어지지 않고 있는 실정이다.
Microarray 기술은 DNA probe를 이용한 hybridization 방법을 통해서 대규모(large-scale) 유전자 발현 양상을 분석할 수 있는 중요한 연구 툴로서 제안되어 왔으며, 1990년대 중반 array 기술이 소개된 이후 많은 연구 기관에서 신뢰성 있는 연구방법으로 사용되어져 왔다[3]. 그러나 지금까지 대부분의 DNA chip을 이용한 microarray 연구는 인간의 생명현상에 관여하는 유전자의 기능, 조절 및 작용 기전을 밝힘으로써 생명현상의 규명과 난치병 치료에 응용하려는 시도로써 이루어지고 있다[4]. 그리고 최근 전장유전체연관성 분석연구(genome-wide association study, GWAS) 또한 노화[5], 당뇨[6], 비만[7] 등과 같은 질환을 중심으로 시도되고 있으며, 운동수행능력 후보 유전자 도출을 위한 일부 연구[8-10]가 진행 중이기는 하나 아직 그 결과는 보고되어지지 않고 있다. 지금까지 보고된 스포츠과학 분야에서의 microarray 연구결과들을 살펴보면 특정 운동에 대한 관련 유전자의 표현형을 밝히기 위한 일부 연구결과[11-16]와 특정 종목 운동선수들을 대상으로 한 다양한 코호트 중심의 연구결과들이 보고되었을 뿐[17], 운동수행능력 차이에 따른 대규모 유전자 발현양상 분석을 통한 우수한 운동수행력 관련 유전자군 발굴에 대한 연구는 아직까지 이루어지지 않고 있는 실정이다.
따라서 본 연구는 저항성 운동수행능력 관련 후보 유전자군을 도출하기 위하여 흰쥐를 대상으로 8주간의 저항성 사다리 운동을 실시한 후 BeadChip을 이용한 BeadArray로 대규모 유전자 발현양상 분석을 통해서 저항성 운동수행능력 관련 후보 유전자군을 제안하고자 한다.
연구 방법1. 연구 대상실험동물은(주)샘타코에서 10주령된 Sprague-Dawley계 수컷 흰쥐 96두를 분양받은 후 통제군(control group, CON, n=16)과 저항성 운동군(resistance exercise training group, RT, n=80)으로 구분하였다. 통제군은 다시 4주차 통제군(CON4, n= 8)과 8주차 통제군(CON8, n= 8)으로 분류하였고, 운동군은 주차별로 기록된 저항성 운동수행능력 검사 결과를 기초로 순위를 정한 후, 상위와 하위 20%에 해당하는 동물을 선정하여 각각 운동 우수군(high performance group, H-RT, 391.36±13.93 g, n=16)과 운동 열세군(low performance group, L-RT, 395.69±10.86 g, n=16)으로 분류하였다. 운동군은 다시 4주차군(H-RT4, n=8; L-RT4, n = 8)과 8주차군(H-RT8, n=8; L-RT8, n=8)으로 구분하였다. 모든 동물에게 일주일간의 환경 적응기간과 운동군은 추가로 일주일 간의 운동 적응기간을 제공하였다. 본 연구에서 제외된 실험동물은 추후 실험에 연구대상으로 제공되었으며, 이상 모든 실험과정은 H대학교 동물실험윤리 위원회의 심의를 받아 진행하였다.
2. 실험동물 처치실험동물은 H대학교 의과대학 동물실험실에서 스틸 케이지(steel cage)를 이용하여 3마리씩 사육하였다. 사육실의 온도는 22±1℃, 습도는 상대습도의 50 ±2%로 일정하도록 조절하였으며, 12시간(08:00-20:00)을 주기로 명암을 조절하였다. 실험동물은 시판되는 고형사료(단백질 22.5%, 지방 3.5%, 저섬유 7.0%, 회분 9.0%, 칼슘 0.7%, 인 0.5%; 삼양사)와 고압 증기로 멸균한 물을 충분히 공급하였다.
실험 시료로써의 조직 채취는 운동 4주 후(16주령)와 8주 후(20주령) 2회 실시하였으며, 저항성 운동과 고형사료는 조직 채취 하루 전에 중단하고 물만 공급하였다. 조직 채취는 zoletil (50 mg/kg)과 rompun (5-10 mg/kg)을 5:1 비율로 혼합하여 복강 내 주입시켜 마취시킨 후 사다리 운동에서 주동근[18]으로 작용하는 장무지굴근(flexor hallucis longus, FHL)의 양측 동일한 부위를 적출하였다.
3. 저항성 운동 프로그램저항성 운동은 동물 꼬리에 부하를 달고 자체 제작한 동물용 사다리(100×12 cm, 2 cm grid, 85° incline)를 오르는 저항성 사다리 운동[18-20]을 변형하여 실시하였다. 최초 부하 없이 사다리를 오르는 운동을 일주일간 실시하여 운동에 대한 적응기간을 가진 후, 꼬리에 부하를 달고 사다리를 오르는 운동을 주당 3일, 10회/일, 총 8주간 점증적 과부하하에서 실시하였다. 저항성 부하는 최대중량의 50% (최초 체중의 50%)부터 시작하여 매회 처음 지정된 무게에서 15%씩 부하를 증가시켰으며, 반복 간 2분간의 휴식 시간을 가졌다. 만약 세트가 끝나기 전에 증량한 부하로 사다리를 오르는 운동 수행이 불가능하게 되면 바로 전 시점의 성공했던 중량으로 세트를 완료하였으며, 더 이상 운동을 할 수 없게 되기 전 시점의 중량을 최대중량으로 기록하였다. 저항성 운동수행능력 평가는 주차별로 기록된 최대중량 무게를 이용하여 실시하였다.
4. 자료처리모든 집단 간 운동 전후에 따른 저항성 운동수행능력의 변화에 대한 분석은 SPSS Ver. 18.0 for Windows 프로그램을 이용하였으며, 모든 측정치에 대한 평균(M)과 표준편차(SD)를 산출하였다. 저항성 운동집단 간(우수군, 열세군) 주차별 저항성 운동수행능력 변화에 대한 차이 검증은 반복측정에 의한 변량분석법(ANOVA with repeated measurements) 을 적용하였고, 상호작용 효과가 유의하게 나타난 결과는 대응 표본 t-검정(paired samples t-test)을 이용하여 집단 간 변화를 비교 검정하였으며, 유의수준은 α = 0.05 수준으로 설정하였다.
5. 분자 생화학적 분석방법1) Total RNA 분리액화질소로 동결된 골격근을 최대한 분쇄시킨 후, Tri Reagent (Molecular Research Center, Inc.)을 첨가하여 상온에서 균질화 시켰으며, chloroform (Sigma)과 isopropanol (Sigma), ethanol을 이용하여 RNA의 추출과 침전, 세척 과정을 거쳐 RNA pellet을 얻었다. 건조된 pellet에 nuclease-free water를 첨가하여 total RNA를 분리한 후 spectrophotometer (NanoDrop)를 이용하여 흡광도와 정제도를 측정하였다. 정제된 total RNA는 Chip 분석과 qPCR에 쓰일 때까지 -70℃에 냉동 보관하였다.
2) BeadArray 분석BeadArray 분석을 위해서 Illumina사의 whole-genome expression인 RatRef-12 Expression BeadChip을 사용하였다. Illumina Expression Direct Hybridization 실험방법은 reverse transcription step을 통해 first와 second strand를 합성하고 그것을 주형으로 하여 in vitro transcription (IVT) amplification을 통해 nucleotide에 biotin을 incorporate 시킨후, 만들어진 probe를 array에 hybridization한 뒤 washing과 streptavidin-Cy3의 staining을 거쳐서 Beadstudio software를 통해서 측정된 값을 분석하는 방법이다. RNA 증폭(amplification)과 표지(labeling), 혼성(hybridization), 세척(washing) 과정을 위해서 Illumina Total Prep RNA Amplification Kit (Ambion), Illumina Gene Expression System Wash E1BC Buffer and Components, Illumina Gene Expression System High Temp Wash Buffer를 사용하였다.
3) 발현 데이터 분석BeadArray data는 Illumina사에서 제공되는 프로그램인 Beadstudio software를 이용하여 normalization과 filtering 등 전처리 과정을 거친 후, 각 유전자의 signal value를 log2로 transformation한 후, 각 유전자별 평균 발현 값[A={log2(control)+log2(condition)}/2]과 control과 condition과의 유전자 발현의 차이 값{M=log2(condition)-log2(reference)}을 이용하여 분석하였다. BeadArray data 중 해당 유전자가 유의하게 발현된 것으로 판별하는 기준은 Detection p-value가 .01 이하인 유전자들 중 M-value가 1.0 이상이고 DiffScore가 30 이상인 유전자만을 선택하여 데이터로 활용하였다. 그리고 hierarchical cluster와 tree view (Eisen; http://rana.lbl.gov/EisenSoftware.htm)를 이용하여 운동집단별과 운동 기간별로 나누어 유전자의 발현양상을 군집분석하였다.
4) Real-time quantitative PCR (qPCR)BeadArray data 검증을 위해서 SYBR Green (Invitrogen)을 이용한 qPCR을 실시하였다. BeadArray data 분석 결과를 기초로 저항성 운동 후 발현의 증가를 보인 특정 유전자 3종(Ahsg, F2, Itih4)과 선행연구에서 보고된 저항성 운동 관련 유전자 2종(Myf6, Ucp3), 총 5종의 유전자를 선택하였다. qPCR에 사용되는 각 유전자의 최적의 primer set는 하버드 대학의 primerBank에서 탐색하여 합성하였으며, 합성한 primer set와 cDNA 샘플을 SYBR Geen supermix와 혼합하여 iCycler를 이용하여 qPCR을 실시하였다. qPCR은 housekeeping gene인 Gapdh와 비교한 상대적인 수치로 표시하였다. 구체적인 qPCR 방법은 iQ SYBR Green Supermix (Bio Rad)를 이용하여 total volume이 50 μL가 되게 만들었다. 또한 qPCR 반응 조건은 denaturation (95℃, 40 sec), annealing (58℃, 40 sec), extension (70℃, 40 sec)으로 하였다. 유전자의 primer sequence는 Table 1과 같다.
연구 결과1. 저항성 운동수행능력저항성 사다리 운동 후 운동 우수군과 열세군의 주차별 저항성 운동수행능력 평가 결과는 Fig. 1에서와 같이 모든 집단에서 주차별로 증가하는 경향을 보였으며, 전반적으로 4주차군(H-RT4, L-RT4)과 8주차군(H-RT8, L-RT8)은 4주차까지 서로 유사하게 증가하는 경향을 보였다(4 weeks groups vs. 8 weeks groups: p>.05).
저항성 운동 4주차군(1195.71±63.47 g vs. 998.57±43.75 g, p<.01)과 8주차군(1464±46.5 g vs. 1286±82.64 g, p<.05) 모두에서 우수군이 열세군에 비하여 유의하게 높은 중량을 들어 올렸으며, 집단 간과 집단 내 변인들 간의 상호작용 효과에서도 4주차군(p<.001)과 8주차군(p<.001) 모두에서 유의한 차이를 보였다.
2. 저항성 운동수행능력 관련 유전자 발현양상 변화4주차(CON4)와 8주차 통제군(CON8)을 기준으로 4주차 저항성 운동군(A: H-RT4, L-RT4)과 8주차 운동군(B: H-RT8, L-RT8)의 BeadArray data에서 M-value를 이용하여 filtering 단계를 거쳐 hierarchical cluster와 tree view를 통하여 전체 유전자(Detection p-value .01 이하)와 집단별 발현의 유의한 차이를 보이는 유전자(M-value 1.0 이상, Diffscore 30 이상)에 대한 군집분석을 실시한 결과는 Fig. 2와 같다.
4주차 운동집단을 살펴보면 유전자의 발현이 증가한 유전자군이 감소한 유전자군에 비하여 상대적으로 높게 나타났음을 알 수 있으며, 특히 저항성 운동 우수군에서 뚜렷한 발현의 증가를 확인하였다. 반면에 8주차 운동군에서는 유전자의 발현이 감소한 유전자군이 증가한 유전자군에 비하여 상대적으로 많은 것을 알 수 있었다.
저항성 운동군 간 유전자의 발현양상을 분석하기 위하여 4주차와 8주차 통제군을 기준으로 각각 4주차(H-RT4, L-RT4)와 8주차 운동군(H-RT8, L-RT8)을 비교하여 분석한 결과는 Table 2와 같다.
4주차군에서 통제군을 기준으로 저항성 운동 우수군(35개 유전자 증가, 6개 유전자 감소)과 열세군(19개 유전자 증가, 7개 유전자 감소) 모두에서 2배 이상(M≥1.0 or M≤-1)의 발현 증가를 보인 유전자는 17개였으며, 감소를 보인 유전자는 4개였다(데이터 미제시). 그리고 24개의 유전자가 열세군에 비하여 우수군에서 2배 이상 발현의 차이가 나타났으며, 이 중 Hp, Ahsg, Ambp, Timp1, Hmox1, Myh6, Mt1a, Igfbp1, Gc, LOC259246, LOC298109, LOC259244, Apoh, MGC72973, Fetub, Itih3, Mup4, Mup5, Slpi, Serpina1 유전자는 발현의 증가가 나타났으며, Dscr1, Atf3, Nr4a3, Tnfrsf12a 유전자는 발현의 감소를 보였다. 반면에 8주차군에서는 통제군을 기준으로 저항성 운동 우수군(7개 유전자 증가, 14개 감소)과 열세군(2개 유전자 증가, 13개 감소) 모두에서 2개 유전자의 발현 증가와 9개 유전자의 발현 감소가 나타났다. 그리고 열세군에 비해 우수군에서 2배 이상의 Ankrd1 유전자 발현 증가와 Egr1 유전자 발현 감소가 나타났다.
3. qPCR 분석 결과qPCR data는 Gapdh와 비교한 상대적인 수치(dCt=gene-Gapdh)를 이용하여 normalization한 Gapdh 대비 상대적인 유전자 발현의 차이값(2n-dCt)을 이용하였으며, BeadArray data는 각 유전자 probe에 대한 average signal 값을 이용하였다. 두 data 모두 4주차와 8주차 통제군에 대한 4주차와 8주차 저항성 운동 우수군과 열세군에서의 발현 변화를 확인함으로써 BeadArray data을 검증하고자 하였으며, 그 결과는 Fig. 3과 같다.
Fig. 3에서와 같이 저항성 운동에 대한 유전자 발현의 증가와 감소의 패턴이 qPCR data와 BeadArray data 간 모든 유전자에서 서로 유사한 양상을 보이는 것을 확인할 수 있었다.
논의Microarray 기술은 대규모 유전자 발현을 동시에 분석함으로써 추후 연구를 위한 잠재적인 표적과 신호전달 체계를 확인하기 위한 유용한 연구방법으로 알려져 왔다[21]. 최근 일부 연구자들은 다양한 손상과 저항성 운동과 같은 근비대 유도를 위한 자극 후 골격근에서 근 손상, 염증, 단백질 합성과 세포수준에서의 기전연구를 위해 관련된 유전자 발현양상을 microarray 기술을 활용하여 성공적으로 수행하였다[11-16]. 그러나 이와 같이 대규모 유전자 발현양상 분석에 효과적인 분석 툴로 알려진 microarray 기술을 활용한 저항성 운동수행능력 관련 유전자 도출을 위한 연구 결과는 아직 보고되지 않고 있는 실정이다. 따라서 본 연구는 whole-genome expression인 RatRet-12 Expression BeadChip (Illumina)을 활용하여 8주간의 저항성 운동 후 저항성 운동 수행 수준 차이에 따른 유전자 발현양상 분석을 통하여 저항성 운동수행능력 관련 유전자를 도출하고자 하였다. 특히 본 연구에서 활용한 BeadChip은 NCBI RefSeq database (Release 16)를 기초로 하여 50 mer의 probe를 갖는 22,523개의 유전자로 구성되어 있으며, 한 유전자 당 30개 이상의 bead를 가지고 있고, 각각 유전자의 발현강도를 사용하는 대신 30개 이상의 유전자에 대한 평균값과 표준오차 값을 이용하여 data를 제공할 뿐만 아니라[3], 한 번 분석으로 각 sequence당 30번 이상의 반복실험 효과를 얻을 수 있기 때문에 결과에 대한 보다 강력한 툴을 제공할 것으로 생각된다.
BeadArray 결과 4주간의 저항성 운동 후 저항성 운동 우수군과 열세군에서 2배 이상의 발현의 증가를 보인 20개 유전자와 감소를 보인 4개의 유전자를 확인하였다.
이 유전자들 중 acute-phase response 관련된 Hp와 Ahsg 유전자는 우수군에서 열세군에 비해 각각 2.11배와 2.36배 발현의 증가가 나타났다. Hp는 antimicrobial, antioxidant, antibacterial activity의 역할과 함께 다양한 acute-phase response를 조절한다. 특히 Hp는 헤모글로빈과 강력한 친화력이 있어 혈장에서 유리 헤모글로빈과 쉽게 결합하여 복합체를 형성하고, 이러한 복합체는 사구체 여과 과정을 거치지 않기 때문에 신장의 산화적 손상과 철분의 손실도 방지하는 것으로 알려졌으며, 항산화제로써 Hp는 immunomodulatory 특성을 보이는 혈장에서 positive acute-phase glycoprotein으로도 작용한다[22,23]. 또한 주로 간세포에서 합성되는 Hp는 IL-6, IL-1β, TNF-α 분비에 의해 생성이 유도 된다[24]. 그리고 Ahsg는 endocytosis, 뇌의 발생과 골 조직 형성 등에 관여하는 것으로 알려졌으며, 특히 골 성장과 재형성의 조절자로서의 작용[25]과 골다공증 관련 유전자로 보고되었다[26].
이 외에도 Anti-inflammatory response 관련 Ambp 유전자(2.75배)는, C21-steroid hormone biosynthesis 관련 Timp1 유전자(2.55배), Heme oxidation 관련 Hmox1 유전자(2.58배), muscle development 관련 Myh6 유전자(2.01배), Nitric oxide mediated signal transduction 관련 Mt1a 유전자(2.01배), regulation of cell growth 관련 Igfbp1 유전자(2.97배), Vitamin D metabolism 관련 Gc 유전자(2.66배)와 기능이 아직 밝혀지지 않은 10개의 유전자가 우수군에서 열세군에 비해 유의하게 발현의 증가가 나타났다.
Ambp는 alpha-1 microglobulin과 bikunin을 분해하는 전구체와 혈장에서 분비되는 복합 당단백을 암호화하고, trypsin inhibitor로서의 기능을 가진다. 그리고 Timp1은 금속단백분해효소 억제제(metalloproteinase inhibitor)로 작용하며, 성장인자로서 세포 분화(differentiation), 세포 이동(migration), 세포사(cell death)를 조절하고 CD63과 Itgb1을 통해서 세포 신호 전달의 역할을 하는 것으로 알려졌다[27]. 한편 Popovici et al. [28]은 Igfbp1 mRNA와 단백질 모두 저산소증 상황에서 발현의 증가를 보고하였으며, Leu 와 George [29]는 Igfbp1을 세포 사멸에서 p53/Bak-dependent pathway의 negative regulator로 보고하였다. 그리고 Gc는 간에서 합성되고 혈장 내 주요 vitamin D-binding protein으로 체내 비타민 D 대사에 관여하며[30], 비타민 D와 혈장 대 사물과 결합되어 표적 조직으로 이들을 운반하는 기능을 가지고 있다. 또한 Gc 단백질은 손상과 염증에 관여하는 것으로도 알려졌다[31]. 그리고 Mehdi et al. [32]은 ApoH cDNA를 간암 세포 주에서 복제 분리하여 염증성 메디아에서 배양시킨 후 ApoH mRNA의 발현의 감소를 보고하고, ApoH를 negative acute-phase protein으로 설명하였다. 또한 ApoH는 지단백질 대사, 혈액응고(coagulation), 항인지질 자가항체(antiphospholipid autoantibodies) [33]의 생성을 포함한 다양한 생리학적 pathways에 관여하는 것으로도 알려졌다.
Oh와 Oh [20]의 연구에서도 본 연구결과에서와 같이 저항성 운동 후 Hp, Ahsg, Ambp, Timp1, Ifgbp1, Gc, ApoH, Mup4, Mup5, Serpina1 유전자의 발현의 증가를 보고하고 이들 유전자를 저항성 운동 관련 후보 유전자로 제안한 바 있다.
한편 4주간의 저항성 운동으로 우수군에서 열세군에 비해 Dscr1 (calcium-mediated signaling), Atf3 (Gluconeogenesis), Nr4a3 (regulation of transcription), Tnfrsf12a 유전자의 유의한 감소가 나타났다. 그리고 8주 간의 저항성 운동에서는 아직 기능이 밝혀지지 않은 Ankrd1 유전자만이 열세군에 비해 우수군에서 3.36배 증가하였고, 학습/기억과 관련된 Egr1 유전자에서 열세군에 비해 2.85배 감소가 나타났다.
결론적으로 본 연구는 8주간의 저항성 사다리 운동 후 운동 수행 수준 차이에 따라 발현의 증가를 보인 유전자 21개와 감소를 보인 유전자 5개를 저항성 운동수행능력 관련 유전자로 제안하며, 향후 이들 유전자들을 표적으로 생물학적, 기능적 기전분석 연구를 위한 기초자료를 제공할 것으로 기대한다.
그러나 본 연구에서는 다음과 같은 연구의 제한점이 있다. 첫째, microarray 분석 기술 툴은 기본적으로 대규모 유전자 발현을 분석을 통하여 발현의 차이를 보이는 유전자군을 중심으로 gene ontology 분석을 활용한 유전자들 간의 생물학적/기능학적 pathway를 밝히는 것이 주요하지만, 본 연구에서는 통계적으로 발현의 차이를 보인 후보 유전자군의 수가 제한적이었기 때문에 유전들 간의 생물학적, 기능학적 상호관계를 분석할 수 없었다. 둘째, 본 연구에서 동물에게 적용된 저항성 사다리 운동은 최초 점증적인 과부하하에서 저항성 운동프로그램을 계획하였으나, 운동 우수군과 열세군 간의 운동수행능력 차이가 발생했음에도 불구하고 운동 4주차 이후 8주차로 진행되는 과정에서 보다 적극적인 과부하 유도가 이루어지지 않은 것으로 보인다. BeadArray data와 qPCR data 모두에서 운동 4주 후 유전자 발현의 증가를 보였으나 4주 이후 8주차에서는 오히려 발현의 감소하는 경향을 확인할 수 있었다.
이와 같은 제한점에도 불구하고 본 연구는 microarray 기술을 이용하여 저항성 운동수행능력 관련 유전자를 도출하려는 최초의 시도로써 연구의 의의가 있으며, 스포츠과학 분야에서 엘리트 운동선수들의 특정 수행능력 관련 유전자 기능분석 연구를 위한 기초 자료를 제공할 것으로 생각된다.
Fig. 1.Fig. 1.Changes of the maximum lifting weight. Groups, H-RT, L-RT; Conditions, 1 week to 8 weeks. Values are mean±SD. H, high performance group; L, low performance group. *p<.05 H-RT4 vs. L-RT4; † p<.05 H-RT8 vs. L-RT8. 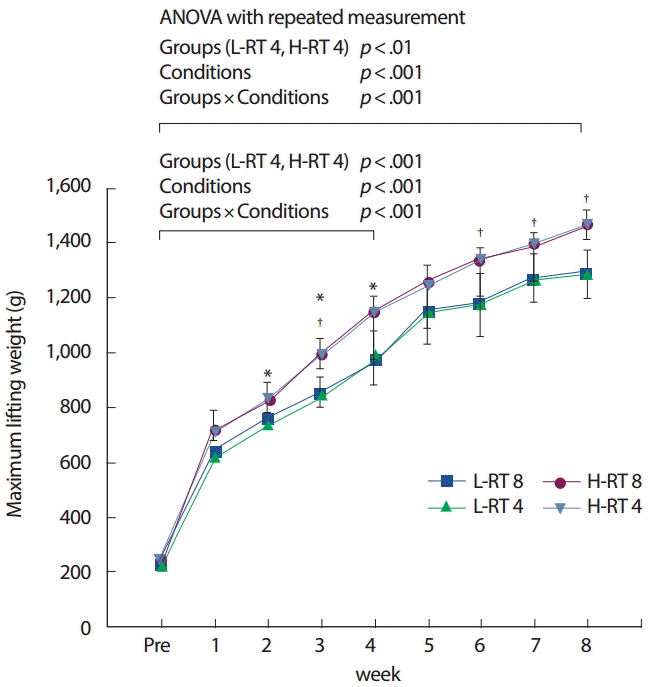
Fig. 2.Fig. 2.Hierarchical clustering: whole data vs. significant data of resistance training groups. Expressed genes of resistance trained groups in contrast with control group; hierarchical clustering data of 4 weeks groups (A) and 8 weeks groups (B). BeadArray data represents the relative difference in gene expression of resistance training groups compared to control groups. Increased genes are shown in progressively brighter shades of red, and decreased genes are shown in progressively brighter shades of green. Values unit are expressed as fold. 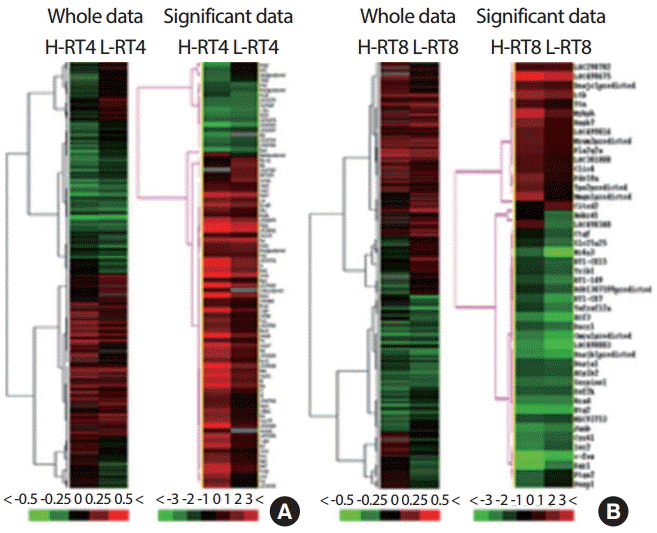
Fig. 3.Fig. 3.Comparison of qPCR data with BeadArray data. The mount of mRNA is shown relative to Gapdh expression. Both qPCR and BeadArray results showed concordant directional as well as proportional changes. Values are mean±SD (aPCR data). H, high performance group; L, low performance group. 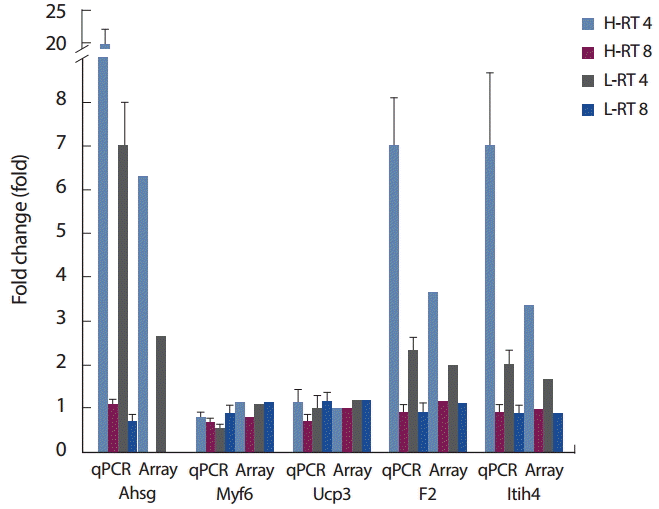
Table 1.Primer sequences for amplification of qPCR Table 2.Expressed genes of resistance trained group in contrasts with 4- and 8-weeks control groups Differential expression of genes in the rat skeletal muscle after 4- and 8-weeks of resistance ladder exercise as revealed by BeadArray analysis. An expression ratio (exercise group vs. control group) of ≥1 or ≤-1 was considered to reflect a significant difference between the two groups. Bold, statistically significant (M>1 or M<-1 (fold >2)). Symbol, unigene symbol; H, high performance group; L, low performance group; C, control group. REFERENCES1. Bray MS, Hagberg JM, Perusse L, Rankinen T, Roth SM, et al. The human gene map for performance and health-related fitness phenotypes: the 2006-2007 update. Medicine and Science in Sports and Exercise. 2009;41(1):35-73.
2. Ahmetov II, Fedotovskaya ON. Current progress in Sports genomics. Advances in Clinical Chemistry. 2015;70:247-314.
3. Wong WC, Loh M, Eisenhaber F. On the necessity of different statistical treatment for Illumina BeadChip and Affymetrix GenChip data its significance for biological interpretation. Biology Direct. 2008;3(1):23.
4. Boenning DA, Kalfoglou AL. The future of clinical research: the role of genomics, informatics, and evidence-based medicine. Journal of Investigative Medicine. 2001;49(1):10-11.
5. Klein RJ, Zeiss C, Chew EY, Tsai JY, Sackler RS, et al. Complement factor H polymorphism in age-relaed macular degeneration. Science. 2005;308(5720):385-389.
6. McCarthy MI, Zeggini E. Genome-wide association studies in type 2 diabetes. Current Diabetes Reports. 2009;9(2):164-171.
7. Frayling TM, Timpson NJ, Weedon MN, Zeggini E, Freathy RM, et al. A common variant in the FTO gene is associated with body mass index and predisposes to childhood and adult obesity. Science. 2007;316(5826):889-894.
8. Wang G, Fuku N, Padmanabhan P, Mikami E, Tanaka M, et al. Genome-wide association study (GWAS) of elite sprinters of West African Ancestry. Paper presented at the XXXII World Congress of Sports Medicine. 2012a;Rome; Italy.
9. Wang G, Fuku N, Padmanabhan P, Mikami E, Tanaka M, et al. Genome- wide association study approach in elite Jamaican athletes to identify common genetic variations associated with world class athlete status. Paper presented at the Glasgow Poyomics Launch Symposium. 2012b;Glasguw, UK.
10. Wang G, Fuku N, Padmanabhan P, Mikami E, Tanaka M, et al. Genome- wide association study of world class athlete status using elite Jamaican sprinters. Paper presented at the Malawi-Liverpool-Wellcome Trust Clinical Research Programme. 2013;Glasguw, UK.
11. Barash IA, Mathew L, Ryan AF, Chen J, Lieber RL. Rapid muscle-specific gene expression changes after a single bout of eccentric contractions in the mouse. American Journal of Physiology Cell Physiology. 2004;286(2):C355-364.
12. Carson JA, Nettleton D, Reecy JM. Differential gene expression in the rat soleus muscle during early work overload-induced hypertrophy. FASEB Journal. 2002;16(2):207-209.
13. Chen YW, Hubal MJ, Hoffman EP, Thompson PD, Clarkson PM. Molecular responses of human muscle to eccentric exercise. Journal of Applied Physiology. 2003;95(6):2485-2494.
14. Chen YW, Nader GA, Baar KR, Fedele MJ, Hoffman EP, et al. Response of rat muscle to acute resistance exercise defined by transcriptional and translational profiling. The Journal of Physiology. 2002;545(1):27-41.
15. Roth SM, Ferrell RE, Peters DG, Metter EJ, Hurley BF, et al. Influence of age, sex, and strength training on human muscle gene expression determined by microarray. Physiological Genomics. 2002;10(3):181-190.
16. Zambon AC, Mcdearmon EL, Salomonis N, Vranizan KM, Johansen KL, et al. Time- and exercise-dependent gene regulation in human skeletal muscle. Genome Biology. 2003;4(10):R61.
17. Pitsiladis Y, Wang G, Wolfarth B, Scott R, Fuku N, et al. Genomics of elite sporting performance: what little we know and necessary advances. British Journal of Sports Medicine. 2013;47(9):550-555.
18. Hornberger TA, Farrar RP. Physiological hypertrophy of the FHL muscle following 8 weeks of progressive resistance exercise in the rat. Canadian Journal of Applied Physiology. 2004;29(1):16-31.
19. Oh SL, Kim HJ, Seong JK, Kim YA, Song W. Effects of acute and chronic resistance exercise training on IL-15 expression in rat skeletal muscle. International Journal of Applied Sports Sciences. 2013;25(2):85-90.
20. Oh SL, Oh SD. Effects of resistance training on skeletal muscle gene expression in rats: a BeadArray Analysis. Journal of Life Science. 2013;23(1):116-124.
21. Maher AC, Fu MH, Isfort RJ, Varbanov AR, Qu XA, et al. Sex differences in global mRNA content of human skeletal muscle. PLoS One. 2009;4(7):e6335.
22. Langlois MR, Delanghe JR. Biological and clinical significance of haptoglobin polymorphism in humans. Clinical Chemistry. 1996;42(10):1589-1600.
23. Melamed-Frank M, Lache O, Enav BI, Szafranek T, Levy NS, et al. Structure–function analysis of the antioxidant properties of haptoglobin. Blood. 2001;98(13):3693-3698.
24. Van Vlierberghe H, Langlois M, Delanghe J. Haptoglobin polymorphisms and iron homeostasis in health and in disease. Clinica Chimica Acta. 2004;345(1-2):35-42.
25. Kalabay L, Cseh K, Pajor A, Baranyi E, Csakany GM, et al. Correlation of maternal serum fetuin/alpha2-HS-glycoprotein concentration with maternal insulin resistance and anthropometric parameters of neonates in normal pregnancy and gestational diabetes. European Journal of Endocrinology. 2002;147(2):243-248.
26. Audi L, Garcia-Ramirez M, Carrascosa A. Genetic determinants of bone mass. Hormone Research in Paediatrics. 1999;51(3):105-123.
27. Chesler L, Golde DW, Bersch N, Johnson MD. Metalloproteinase inhibition and erythroid potentiation are independent activities of tissue inhibitor of metalloproteinases-1. Blood. 1995;86(12):4506-4515.
28. Popovici RM, Lu N, Bhatia S, Faessen GH, Giaccia AJ, et al. Hypoxia regulates insulin-like growth factor-binding protein 1 in human fetal hepatocytes in primary culture: suggestive molecular mechanisms for in utero fetal growth restriction caused by uteroplacental insufficiency. The Journal of Clinical Endocrinology, Metabolism. 2001;86(6):2653-2659.
29. Leu JI, George DL. Hepatic Igfbp1 is a prosurvival factor that binds to Bak, protects the liver from apoptosis, and antagonizes the proapoptotic action of p53 at mitochondria. Genes, Development. 2007;21(23):3095-3109.
30. Daiger SP, Schanfield MS, Caballi-Sforza LL. Group-specific component (Gc) proteins bind vitamin D and 25-hydroxyvitamin D. Proceedings of the National Academy of Sciences. 1975;72(6):2076-2080.
31. Gomme PT, Bertolini J. Therapeutic potential of vitamin D-binding protein. Trends in Biotechnology. 2004;22(7):340-345.
|
|
|||||||||||||||||||||||||||||||||||||||