운동과 장내 미생물: 연구방법의 발전과 연구동향
Exercise and Gut Microbiome: Trends and Advances in Research Methods
Article information
Trans Abstract
PURPOSE
To suggest future research directions and current research trends based on representative studies of existing exercises and the gut microbiome. In addition, we reviewed methodologies to study the relationship between exercise and the gut microbiome.
METHODS
The research methodologies and results were integrated through a literature review of the latest “exercise and gut microbiome” studies and a narrative review.
RESULTS
Although exercise is indirectly related to the gut microbiome or immunity, evidence for a direct effect is still lacking. However, with the recent discovery of gut microbiomes that can help improve exercise performance, it is clear that exercise can positively alter the gut microbiome.
CONCLUSIONS
Strong evidence suggests that regular moderate exercise improves overall immune function and lowers the incidence of inflammation-related disease. In addition, certain microorganisms affect exercise performance.
서 론
인간은 피부(skin)와 구강(oral cavity) 및 구강에서 항문으로 이어지는 소화기 계통(gastrointestinal tract), 여성의 생식기인 질(vagina) 등에 존재하는 다양한 미생물(microorganism)과 공생(symbiosis)하며 상호작용한다[1]. 즉, 미생물의 숙주(host)인 인간은 미생물에 의해 영양, 대사기능(metabolism) 그리고 면역계와 상피세포(epithelial cell) 성숙에 영향을 받는다[2].
건강을 유지하기 위해서는 신체 전반에 걸쳐 “미생물 균형 환경(eubiosis)”을 유지하는 것이 중요하다[3]. 특히, 장내 미생물 환경(gut microbiome)은 음식물 소화에 도움을 줄 뿐만 아니라 최근 다양한 연구를 통해 면역과 대사활동 등 건강한 신체를 유지하기 위한 중요한 역할을 담당하는 것으로 알려져 있다[3]. 장내 “미생물 균형이 무너진 상태(dysbiosis)”는 비만[4], 제2형 당뇨[5], 염증성 장 질환(inflammatory bowel disease) [6] 그리고 대장암(colon cancer)과 관련이 있는 것으로 잘 알려져 있으며[7], 심지어 자폐증(autism)과도 관련이 있는 것으로 알려져 있다[8]. 이러한 질병과의 연관성은 건강의 유지 및 개선에 있어 장내 eubiosis의 유지가 중요한 목표가 될 수 있음을 시사한다.
다양한 질병과의 관련성에 근거하여 장내 eubiosis를 유지하는 것은 건강관리를 위해 중요하다고 생각된다. 규칙적인 신체 활동과 적당한 운동은 전반적인 면역기능을 향상시키고 염증 관련 질환의 유병률을 감소시키는 효과가 있는 것으로 알려져 있기 때문에[9,10], 신체 활동 및 운동과 장내 미생물과의 유의미한 관련성을 예측할 수 있다.
운동은 일생에 걸친 면역기능의 유지에 도움을 주고 선천 면역의 강화를 통하여 외부 항원에 대한 방어력을 유지하는 데 도움을 줄 수 있는 것으로 잘 알려져 있다[11,12]. 그러나 규칙적인 운동이 장에 미치는 영향에 대한 해석은 여전히 완벽하지 못하여 “운동-장내 미생물”과의 직접적 인과성을 증명하기가 쉽지 않은 것이 현실이다. 최근 Jeffrey A. Woods 교수의 인터뷰에서도 현재까지 면역학적인 관점에서 운동이 장내 미생물이나 장 면역에 미치는 영향에 대해서는 연구가 더욱 활발히 이루어져야 한다고 언급하기도 하였다[13]. 따라서 우리는 운동이 장내미생물 환경에 미치는 영향에 관련한 대표적인 연구를 바탕으로 심층적으로 검토하고자 하였다. 이를 통해 운동이 장내미생물 환경에 미칠 수 있는 가장 직접적인 효과가 무엇인지 파악하고, 더 나아가 후속연구에서 다루어져야 할 연구와 기존 연구 간의 간극(missing link)을 파악하는 것이 본 검토의 궁극적인 목적이다. 또한, 이러한 목적에 접근하기 이전에 먼저 건강과 질병에 대한 장내 미생물의 영향에 대한 이해와 함께 운동과 장내 미생물 연구를 가능하게 하는 방법론에 대한 검토를 실시하고자 한다.
장내 미생물 연구 방법의 발전
1. 미생물 군 유전체(microbiome) 연구 방법의 발전
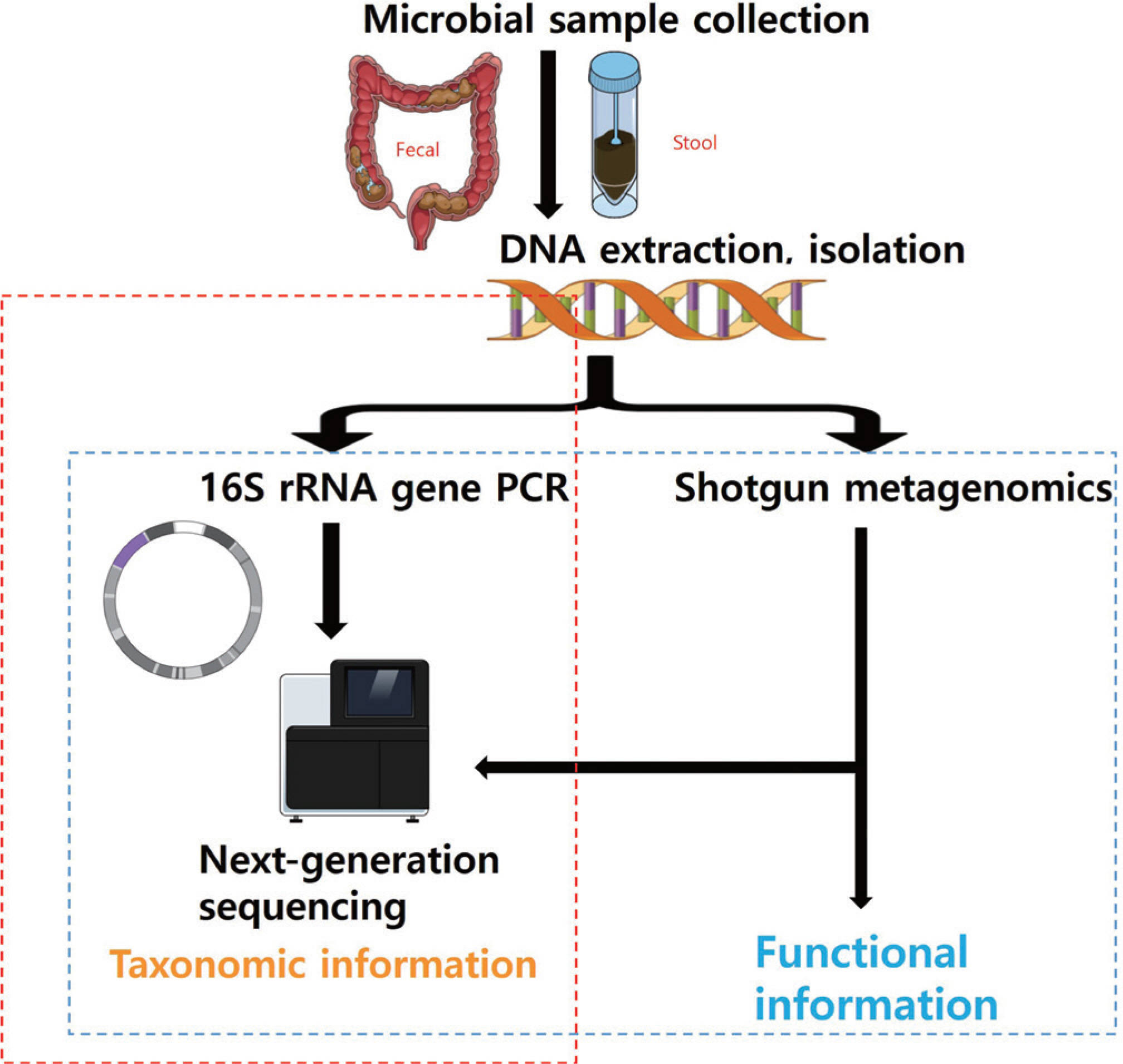
초기 장내 미생물 연구는 주로 배양, 염색 및 현미경 검사를 기반으로 하였다. 하지만 배지 조성과 같은 일반적인 배양 조건은 호기성 미생물을 선호하므로 혐기성 미생물을 효과적으로 배양하거나 연구할 수 없다는 단점이 있었다[14]. 이러한 단점은 차세대 시퀀싱(next gener-ation sequencing, NGS)이 개발되고 상용화됨으로써 획기적으로 보완되었고[15], 이로 인해 인체 미생물 군집 관련 연구가 박차를 가하게 되었다[16,17]. NGS의 지속적인 기술적 진보와 함께 대용량 데이터를 처리할 수 있는 생물정보학(bioinformatics)적 분석법이 비약적 발전을 이룩함으로써 인간뿐만 아니라 동물, 식물 그리고 미생물 등 다양한 생명체의 유전체분석이 이루어지기 시작하였다[15]. 또한, RNA-seq으로 불리는 전장전사체분석(whole transcriptome shotgun sequencing, WTSS), 전장엑솜분석(whole exome sequencing, WES) 그리고 DNA 메틸화분석(methylation sequencing, MeS) 등의 응용분야로 점차 확대되었다. 최근에 장내 미생물 연구는 NGS를 기반으로 하여 마이크로바이옴 메타지놈(microbiome metagenome) 및 메타트랜스크립톰(metatranscriptome) 연구로 그 흐름이 이어지고 있다(Fig. 1).
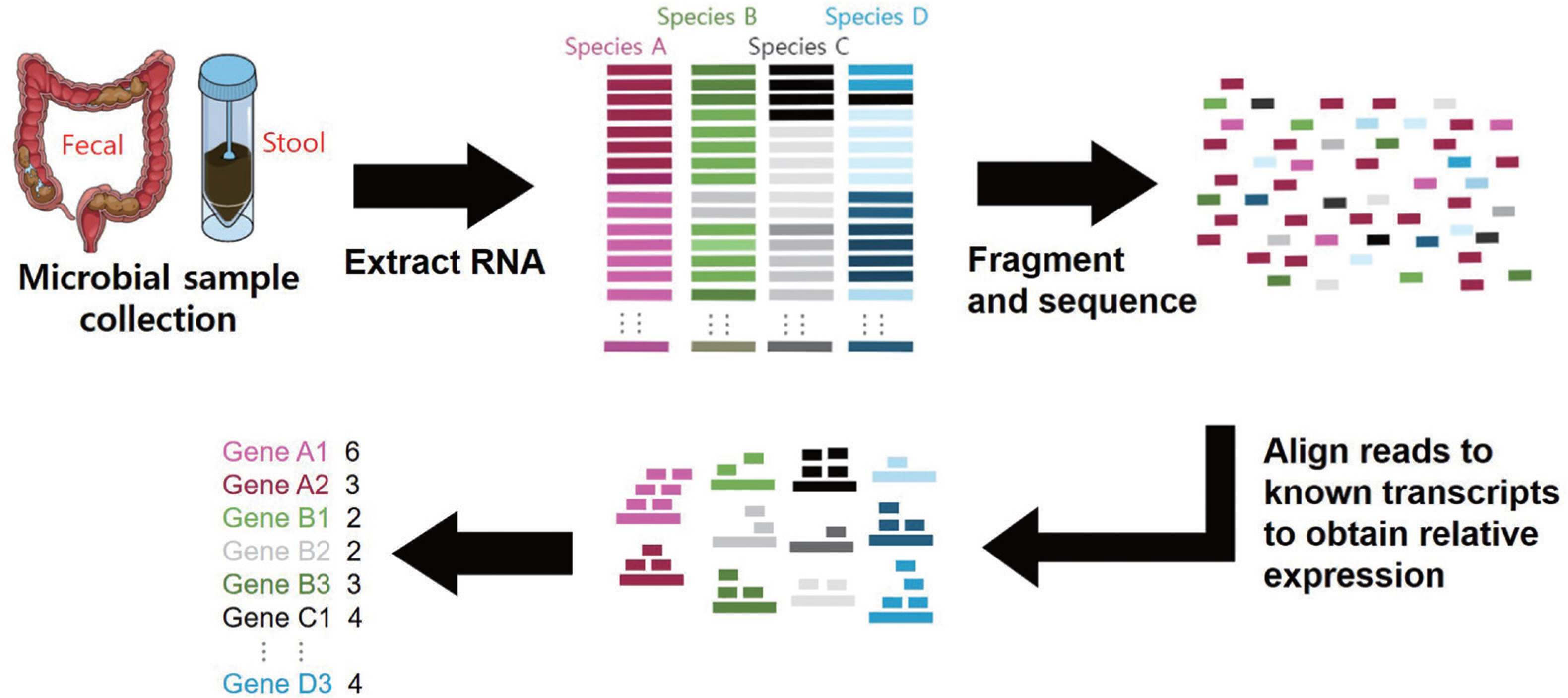
2. 메타지노믹스(metagenomics)와 메타트랜스크립토믹스(metatranscriptomics)
메타지노믹스의 목적은 미생물 군집을 분석하고 그 안의 잠재적인 기능평가도 가능하여 숙주와 미생물 사이의 유기적인 관계를 이해하는 목적이 있다(Fig. 2). 앰플리콘 시퀀싱(amplicon sequencing)은 이러한 목적 중 미생물 군집(community)을 분석하는 데 주요 목적이 있다. 이때 종 동정(species identification)을 위한 표지 유전자(marker gene)만을 선택적으로 증폭한 후 그 증폭 산물들의 염기서열을 분석하게 된다[18]. 따라서 검체 내에서 세균이나 고균(archea)에서 발견되는 원핵생물(prokaryote)의 DNA인 16S rRNA (16S ribosomal RNA) 유전자를 표지유전자로 사용하게 된다[18]. 이러한 절차를 간단하게 설명하면, 바코드서열이 들어있는 프라이머(primer)를 이용하여 16S rRNA 유전자를 PCR (polymerase chain reaction)으로 증폭하고 16S rRNA 데이터베이스인 Greengenes (http://greengenes.lbl.gov), SILVA (https://www.arb-silva.de/) 또는 RDP (http://rdp.cme.msu.edu/)를 이용하여 각각의 염기서열을 대조하여 미생물 종을 알아낸다[19–21]. 세균에 존재하는 16S rRNA 유전자는 그 길이가 적당할 뿐만 아니라 보존영역(conserved region)과 변이영역 (variable region) (V1–V9)을 적절히 포함하고 있다. 또한, 기능 변화에 따른 유전자의 변이 가능성이 낮아 계통 분석과 생태 연구에 적합하다. 이러한 장점들로 앰플리콘 시퀀싱은 특정 환경의 미생물 구성을 적은 비용으로 빠르게 특성화할 수 있다. 16S rRNA의 리보좀 영역을 해독하는 방법은 앞서 언급한 프라이머로 V1– V9의 가운데를 타겟으로 하는 영역들을 증폭하여 해독하는 방법이 가장 널리 쓰이는 방법이다. 특히, 일루미나(Illumina, San Diego, CA, USA) 사의 플랫폼을 이용하여 해독할 경우 짧은 단짝 서열(short read pair end)이 해독되고, 이를 바탕으로 데이터베이스에 존재하는 서열들에 매핑(mapping)하여 미생물의 속(genus)과 종(species)을 구분한다. 다만, 종 수준으로 분류하기 위해서는 여러가지 한계점을 가지고 있다. 이러한 단점을 보완하기 위하여, 일명 산탄총 메타게놈(shotgun metagenome) 해독 방법이 사용되는데 특정 영역을 타겟해서 해독하는 것이 아니라 샘플 내에 있는 genomic DNA를 잘라서 산탄총처럼 랜덤으로 라이브러리를 제작하여 해독을 하는 방법이다. 이는 샘플 내에 존재하는 미생물의 DNA를 해독하는 방법이기 때문에 유전자 영역들이 포함되어 해독이 이루어지게 된다. 따라서 미생물의 기능을 분석하거나 미생물을 종 수준까지 특정하기에 용이한 방법이다. 또한, 프라이머 조합을 이용하여 해독된 16S rRNA 전 영역의 단짝 서열을 조립하여 마치 전체 16S 리보좀을 해독한 것과 같은 효과를 얻을 수 있는 긴 서열(long read) 합성 방법(loop-sequencing) 또한 명확하게 미생물을 종 수준까지 특정하는 데 효과적인 방법이 될 수 있으며, 현재는 Loop Genomics (San Jose, CA, USA)사의 loop-seq 방법이 활용되고 있다[22].
장내 미생물들의 활성이 숙주의 특정 표현형(phenotype)으로 발현되기 위해서는 미생물 군집 내에서의 상대적인 숫자뿐만 아니라 실제 존재하는 절대 수가 큰 영향을 미친다. 건강 유지에 매우 중요한 역할을 담당하는 특정 균주는 종 다양성의 차이로 인해 다양성이 매우 제한적인 환자의 메타지놈보다 상대적으로 월등히 많은 수의 다양한 미생물을 보유하고 있는 건강한 사람의 메타지놈에서 더 높게 나타날 수 있다[23]. 그렇기 때문에 메타지노믹스를 통한 정량 분석은 실제 활성을 갖는 균주와 유전자 그룹을 추론하는 데 한계가 있다. 따라서 실제 유전자가 발현된 메타트랜스크립토믹스 또는 메타프로테오믹스를 활용하는 것이 활성 미생물 그룹 분석에 이점이 있다[24]. 실제로 마이크로바이오타(microbiota)라고 불리우는 미생물 군집의 개인 간의 차이가 커서 질병 특이적인 바이오마커를 발굴하는 데 여전히 많은 어려움을 겪고 있으며, 이를 극복하기 위해 실제 활성을 보이는 미생물 종과 발현되는 유전자 그리고 대사경로를 함께 분석하는 메타트랜스크립토믹스가 각광을 받고 있다[24–26].
운동과 장내 미생물과의 관련성
1. 동물연구에서의 근거
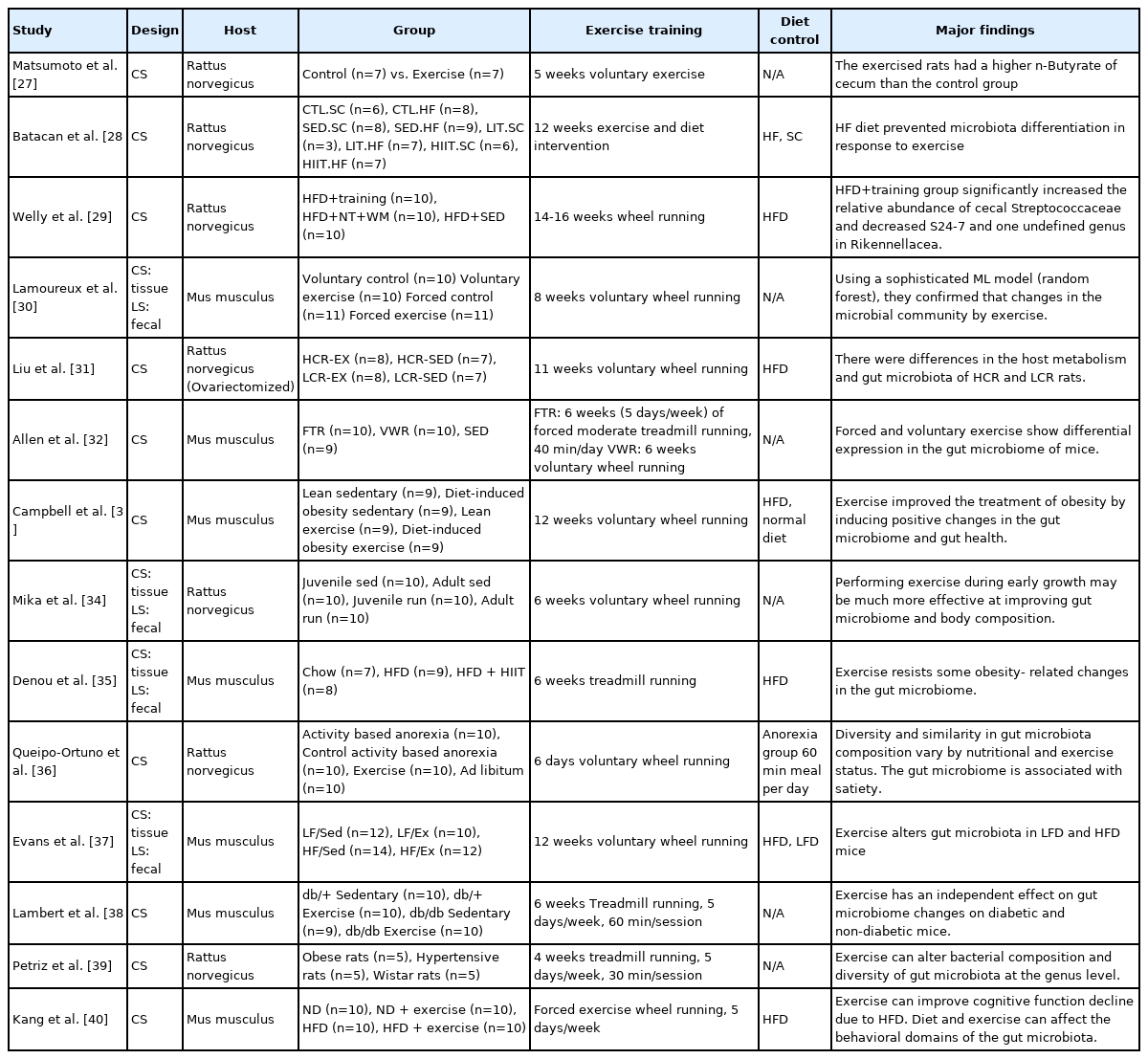
엄격히 통제된 동물 연구는 운동과 장내 미생물 환경과의 관계를 파악하는 데에 있어 실마리를 찾는 데 효과적인 방법이다(Table 1). 특히, 5주간의 운동 훈련을 통해 박테리아의 대사산물인 부티레이트(butyrate)를 증가시키는 것을 최초로 발견하는 데 일조를 하였다[27]. 이러한 이유로 인해 많은 동물 연구가 수행되었고, 동물 연구에서 밝혀진 다양한 연구들을 근거로 운동은 독립적으로 장내 미생물의 구성과 기능적 능력 변화에 영향을 미치는 것으로 파악되고 있다[27–40]. 앞서 언급된 바와 같이 운동은 독립적으로 장내 미생물 환경의 변화를 야기할 수 있고 이는 부티레이트의 증가를 초래하는 것으로 보여진다. 이후에 이루어진 연구에 의해 이러한 결과의 원인은 운동을 통하여 부티레이트를 생성하는 미생물 군집의 상대적인 숫자를 증가시키기 때문으로 확인되었다[37–40]. 식이섬유의 박테리아 발효로 인해 생성되는 단쇄지방산(short chain fatty acids, SCFAs)인 부티레이트는 결장 상피세포(colonic epithelial cell)의 증식을 촉진시키고 장벽(gut barrier)의 건강유지 및 숙주의 면역 시스템과 유전자 발현을 조절하는 것으로 여겨진다[41,42].
운동이 실험동물(대부분 설치류)의 장내 미생물 환경을 어떻게 그리고 어느 정도로 변화시키는지에 대한 포괄적이고 정량적인 결론을 도출하는 데 있어서는 식단과 실험동물의 종(species)과 계통(strain), 동물의 주령 및 사용된 운동방식이 일치하지 않기 때문에 상당히 어려운 것으로 보여진다. 예를 들면 Firmicutes/Bacteriodetes 문(phylum)의 장내 비율이 운동에 의해서 증가한다고 보고되는 경우가 있기도 하지만 [38–40], 다수의 반대 결과도 있기 때문이다[34–37]. 또한, 운동으로 인해 장내미생물의 변화가 일어나지 않는다고 보고한 결과들도 있었다. 따라서, 국내에서 운동생리학 관련 연구자들이 “운동과 장내 미생물” 연구에 이용할 수 있는 가이드라인 또는 프로토콜들을 잘 정립하고 표준화하는 작업을 거친 다음 이를 바탕으로 수행된 연구결과로 토론이 이루어져야 할 것으로 생각된다. 최근 인간의 유산소성 운동을 가장 잘 표현하는 두 가지 방법인 자발적 휠 달리기(voluntary wheel running)와 강제성이 있는 트레드밀 달리기(forced treadmill running)를 실시하였을 때에 장내 미생물 군의 변화 양상이 다르게 나타났다[32]. 이러한 사실은 운동 프로토콜이 실험결과를 좌우할 수 있음을 보여준다.
2. 인간 대상 연구에서의 근거
동물연구에서의 결과가 운동이 장내 미생물 군의 변화에 분명히 영향을 미친다는 것을 과학적으로 입증하고 있지만, 운동으로 유발된 변화가 인체에 유익한 방향으로 작용한다는 것을 명확히 입증하지는 못하였다. 전술한 “동물연구에서의 근거”에서 언급한 바와 같이 여러 연구들에서 표준화되지 않은 연구방법 때문에 이러한 문제가 발생한다. 운동생리학자의 관점에서 기존의 수많은 연구들에 입각하여 건강상의 이점을 득하기 위한 적절한 운동형태, 운동강도 또는 운동빈도에 대한 근거들을 통합적으로 이해하고 있다. 따라서 이러한 이해를 바탕으로 이전에 이루어진 동물연구에서의 발견을 인체에 확대적용하여 운동이 장내 미생물 환경을 변화시키는지 확인하고 추가적으로 건강상의 이점을 득하기 위한 적절한 운동형태, 강도 또는 운동빈도를 제시하는 것은 운동생리학 또는 운동과학에 있어 중요한 목표가 될 것이다.
최근에 인간을 대상으로 이루어진 종단 연구의 결과는 운동에 의한 미생물 군은 체질량지수(body mass index, BMI)에 따라 차등적으로 변경되는 것을 보여주었다[43]. 해당연구에서 장내 Faecalibacterium 종이 정상 대상자(BMI <25)에서 운동에 의해 증가하는 것을 보여주었다. 하지만 비만 피험자는 그렇지 않았다. 또한, Bacteriodetes는 정상 대상자에서 감소하고 비만 대상자(BMI >30)에서는 증가하였다. 이러한 결과들은 운동에 의한 장내 미생물의 변화는 식이와 건강상태에 의해 크게 좌우될 수 있음을 보여주는 결과라고 생각된다. 해당 연구에서 시행된 6주간의 지구성 운동으로 인하여 부티레이트를 생성하는 미생물 군이 정상 대상자에서만 효율적으로 변화하였기 때문에 이러한 추측은 좀 더 명확한 것으로 보여진다. 해당연구에서 가장 중요하게 생각되는 결과는 운동과 함께 증가하는 대부분의 미생물 군과 SCFA는 이후에 수행된 좌식생활로의 전환을 통해 다시 감소하였음을 보여준 것이었다[43]. 이는 운동에 의한 장내 미생물 환경의 긍정적인 변화가 일시적일 뿐만 아니라 가역적임을 보여주는 결과였다(Table 2).
앞서 언급한 연구결과와 마찬가지로 식이가 운동으로 인한 장내 미생물 변화에 주요한 변수가 될 수 있음을 보여준 추가적인 연구결과가 있다[44]. 해당연구에서 유청단백질만을 섭취한 과체중 또는 비만 성인에 비하여 운동단독 또는 운동과 유청단백섭취를 병행한 과체중 또는 비만 성인에서 미생물의 다양성이 증가하는 경향을 보여주었다(운동군은 중강도의 유산소운동을 실시함). 다만, 미생물 군집의 특이적인 변화와 이로 인한 대사체의 변화가 크지 않다는 것 또한 보여주었다. 이러한 결과는 앞서 언급한 연구에서의 결과와 마찬가지로 장내 미생물 변화의 특성상 평소의 식이습관과 대상자의 다양한 BMI 분포에 기인하여 좀 더 특이적인 변화를 감지하지 못하였을 수도 있기 때문으로 사료된다. 이러한 결과를 좀 더 비판적으로 바라보면 장내 미생물 변화를 유도하는 것으로 여러 연구에서 확인된 운동의 효과를 단백질 섭취가 상쇄하였을 수도 있음을 고민할 필요가 있을 것이며, 추후 연구될 가치가 있을 것이다.
지구성 운동이 과체중 여성의 장내 미생물 군집에 영향을 미칠 수 있는지 확인한 연구에서 6주간의 저강도와 중강도 사이의 사이클링을 수행하였을 때에 A. muciniphila 종을 상대적으로 증가시키고 Pro-teobacteria를 감소시키는 것이 확인되었다[45]. 해당 연구의 가장 중요한 발견은 운동에 의해 반응하는 미생물 군집은 예상과 달리 전체 장내 미생물의 절반밖에 되지 않는 것으로 확인된 것이다. 또한, 운동은 과당 및 아미노산 대사와 관련된 미생물 군집의 기능 유전체를 감소시킨 것으로 확인되었다.
3. 인간 대상 연구에서 횡단 연구를 통한 근거
인간의 장내 미생물 군을 형성하는 데 있어 운동의 역할에 대한 근거는 횡단 연구에서 최초로 확인되었다[46]. 해당 연구에서 엘리트 럭비 선수의 장내 미생물 군은 건강한 일반 대조군에 비해 장내 미생물의 알파 다양성(α-diversity, 평균 종 다양성)이 크고 40여 가지의 다른 박테리아 분류군이 상대적으로 많다는 것을 발견하였다. 또한, 엘리트 럭비 선수는 건강한 일반 대조군에 비하여 Bacteroides와 Lactobacillus 종이 적다는 것을 확인하였다[46]. 최근에 활동적인 여성과 좌식 대조군을 비교한 결과 주당 3시간 이상의 운동을 하는 여성이 부티레이트를 생성하는 것으로 알려진 Faecalibacterium prausnitzii, Roseburia hominis 및 Akkermansia muciniphila, F. prausnitzii 와 R. hominis의 수준이 증가해 있는 것을 확인하였다[47]. 특히, A. municiphila는 제지방량 및 대사건강과 관련이 있는 것으로 확인되었다[48].
여러 연구에서 미생물 군집과 대사능력 및 심폐 건강의 관련성을 밝히고자 하는 시도가 이루어졌다. 관련된 연구에서 인간 장내 미생물 총(microbiota)의 두 가지 주요 문인 Bacteriodetesd와 Firmicutes의 비율이 높을수록 최대산소섭취량이 높은 것으로 확인되었다[49]. 유사한 결과로 성인에서 미생물 다양성과 부티레이트를 생성하는 박테리아 분류군의 풍부함(abundance)이 심폐 건강과 양의 상관관계가 있는 것으로 확인되기도 하였다[50]. 또한, 메타지놈 분석을 사용하여 운동 선수가 아미노산 생합성 및 탄수화물 대사뿐만 아니라 더 높은 대변 SCFA 농도를 나타내어 장내 미생물을 변화시켰음을 확인하였다[51].
종단 연구와 횡단 연구는 상호보완적이다. 특정 인구집단을 구분하여 접근하는 것이 과학적으로 명확한 것처럼 보이지만 실상은 그렇지 않은 경우가 많다. 즉, 본 검토에서 확인된 여러 연구들에서 사용된 횡단 디자인은 다양한 통제(식이, 일상활동량, 수면패턴 등)가 불가능하여 다양한 요인들의 영향을 제어할 수 없다는 제한점이 있었다.
최신 연구 동향과 미래 연구 방향
앞서 언급한 여러 동물연구의 근거들과 인간 대상 연구들은 장내 미생물 군집의 변화를 긍정적으로 유발하는 것으로 보여진다. 하지만 출판된 연구들의 대부분은 결론적으로 보고된 결과물이며, 대체로 유의한 결과들만을 보여주고 있기 때문에 보고되지 않은 결과들에 대한 고려가 없는 생존자 편향(survivorship bias)이 존재할 수 있다는 가능성을 무시해서는 안된다. 특히, 본 검토에서 언급한 한 연구에서 엘리트 럭비 선수들의 단백질 섭취가 장내 미생물을 변화시키는 것으로 나타났지만, 횡단 연구의 특성과 맞물려 종속변인인 장내 미생물을 오직 단백질에 의한 종속관계인 것으로 판단하는 우를 범할 수 있다는 사실을 간과해서는 안된다[46]. 이러한 단점을 보완하기 위해서는 동물연구 또는 인체 종단연구에서 이를 교차검증할 필요성이 있다. 결정적으로 지나친 고단백 식이를 실시하였을 때 오히려 장내 미생물 환경과 안정성을 교란시키고 이로 인해 스프린트 및 지구력이 감소할 수 있다는 최근 결과가 보고되고 있어 교차검증의 필요성이 더욱 중요해지고 있다[52]. 앞으로의 연구에서는 장내 미생물 환경의 개선을 위한 최선의 방법을 운동, 휴식 그리고 영양의 최적의 조합을 찾는 방향으로 이루어져야 할 것이다.
최근의 연구에서는 NGS 및 바이오인포매틱스의 비약적인 발전을 바탕으로 엘리트 운동 선수에게서 특이적으로 발견되는 장내 미생물 종을 동정하여 이를 이식하는 방법을 통해 운동 능력을 향상시키고자 하는 노력이 이루어지고 있다[53]. 운동능력은 지속적인 트레이닝을 통해 향상시킬 수는 있지만 유전적 요인은 운동능력을 표현형으로 나타내는 데 매우 중요한 인자일 수 밖에 없다. 따라서 운동능력을 향상시키기 위해 장내 미생물을 발굴하고 특정 미생물 군의 이식을 통해 유전적 한계를 극복하고자 하는 노력이 이루어지고 있는 것이다. 실제로 2015년 보스턴 마라톤에 참가한 선수에서 획득한 대변 샘플에서 Veillonella atypica를 분리동정 해내는 데 성공하였고, 이를 실험동물(C57BL/6 마우스)의 장내에 접종하는 실험을 실시하는 시도를 하였다. 결과는 놀랍게도 실험동물의 지구력의 향상으로 나타났다[53]. 미래의 연구에서는 기존에 이루어지고 있는 운동과 관련된 특정 미생물 군과 종의 분류 작업이 고도화되는 것이 중요할 것이다. 절차적인 어려움이 존재하지만 궁극적으로는 분류된 특정 미생물 종의 인체 적용을 통하여 운동의 치료적 효과를 간접적으로 전달하는지 확인하고, 이를 운동부족으로 인해 발병하는 대사증후군을 가진 대상자에게 적용 가능한지에 대한 연구가 집중적으로 이루어져야 할 것이다.
Notes
이 논문 작성에 있어서 어떠한 조직으로부터 재정을 포함한 일체의 지원을 받지 않았으며, 논문에 영향을 미칠 수 있는 어떠한 관계도 없음을 밝힌다.
AUTHOR CONTRIBUTION
Conceptualization: K Baek, J Kim; Data curation: K Ahn, YJ Ahn; Formal analysis: K Baek; Funding acquisition: K Baek; Methodology: K Baek; Project administration: K Baek; Visualization: K Baek, Y Xiang; Writing - original draft: K Baek, K Ahn, YJ Ahn; Writing - review & ed-iting: K Baek, K Ahn, YJ Ahn.