서 론
연구 방법
1. 연구 대상
2. 연구내용
1) DNA 추출
2) Genome-Wide Association Genotyping
3. 자료처리방법
연구 결과
1. GWAS에 의한 경기력관련 타겟 SNPs 수의 도출
1) 분석대상의 총 SNP 개수
2) QC를 통한 최종 분석대상의 SNP 결정
3) GWAS 결과
Fig. 1.
Fig. 1.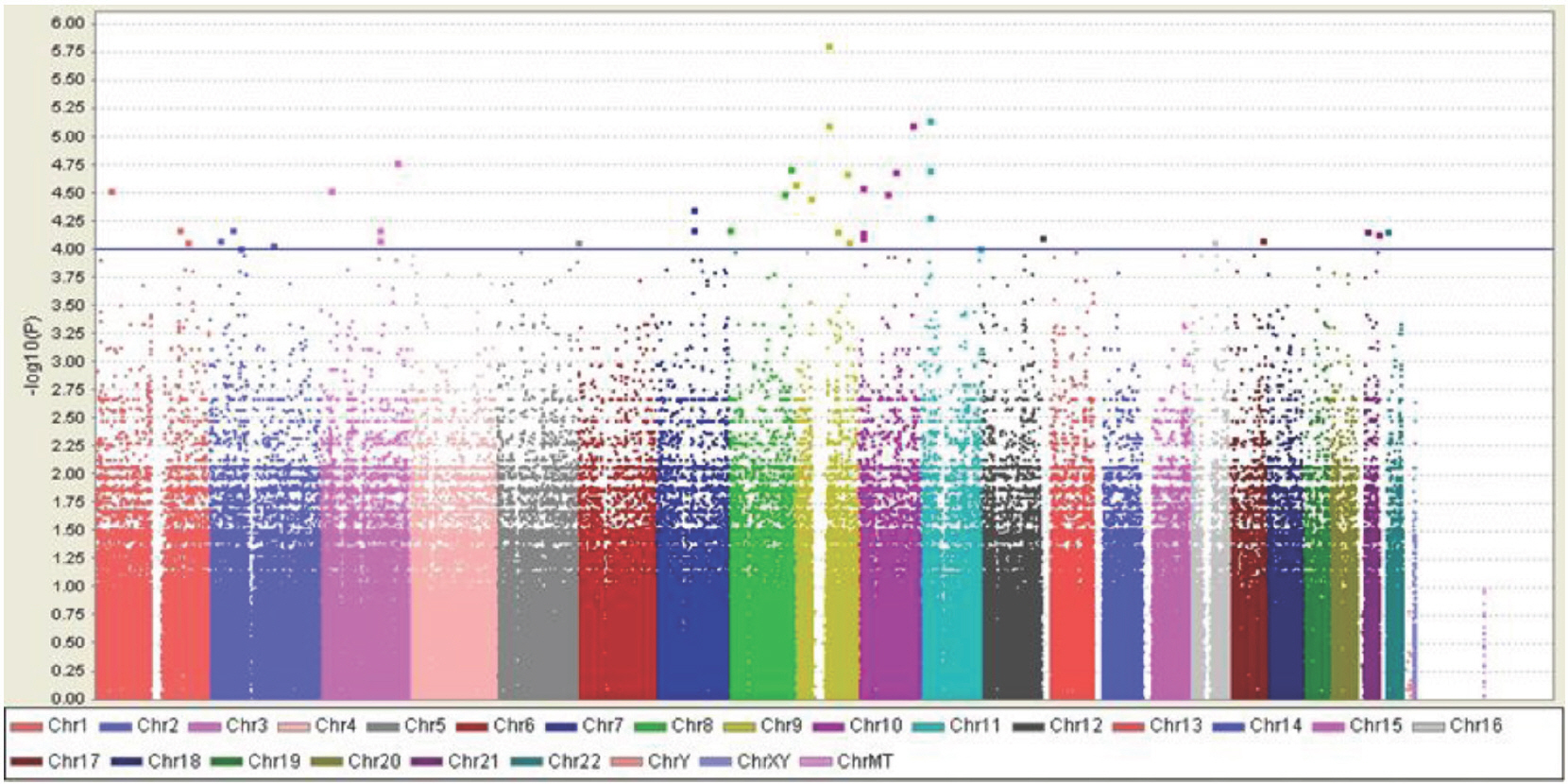
Fig. 2.
Fig. 2.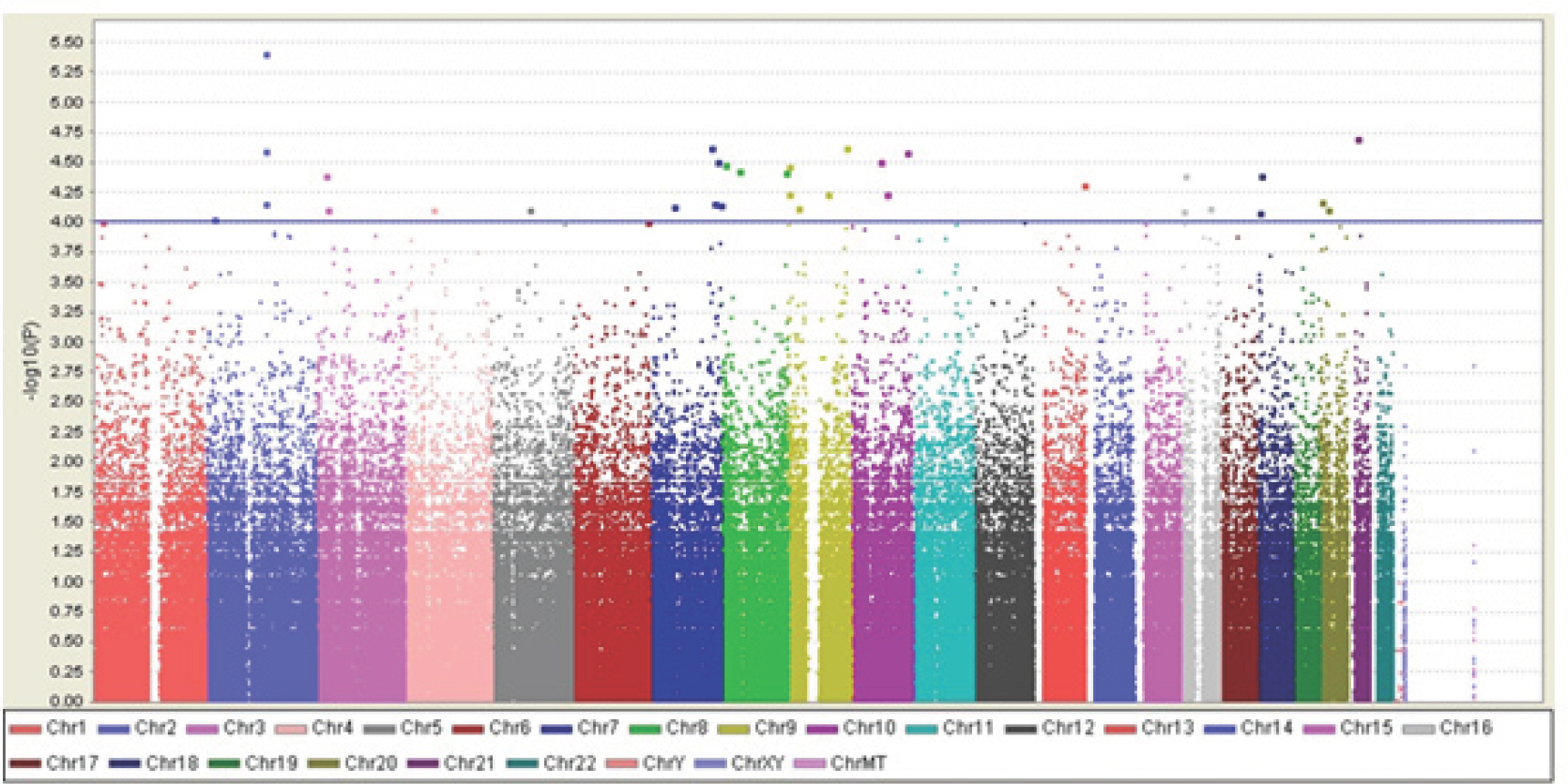
AbstractPURPOSEThe purpose of this study is to analyze SNPs related to performance (maximal strength vs. cardiovascular endurance) of world-class Korean elite athletes using the Genome-Wide Human SNP Array 6.0 and to determine genotypes related to maximal strength and cardiovascular endurance performance that are unique to “ Koreans”.
METHODSUsing the Genome-Wide Human SNP Array 6.0, we analyzed SNPs obtained from 54 world-class Korean elite athletes (Olympic participants/World Championship medalists). The group was divided into the 16 maximal strength group (12 males and 4 females weightlifters who had won medals at the Olympic and World Championships), 12 cardiovascular endurance group (12 males marathon runners who had run within 2 hours and 12 minutes in international competitions), and 26 individuals from the general population (25 males and 1 female). All the obtained SNPs were subjected to quality control (QC), and a total of 867,982 SNPs were analyzed between the marathon runners and general population subjects (641,040 SNPs) and between weightlifters and general population subjects (634,418 SNPs).
RESULTSThe number of SNPs with more than two SNPs within 100 kb, which satisfied the threshold of sample QC and significance level of p<.0001, was 60 in marathoners and 18 in weightlifters compared to the general population subjects. When the genomic features of SNPs of each performance trait were analyzed, the 60 SNPs for cardiovascular endurance with two or more SNPs within 100 kb that met the p<.0001 threshold for significance were distributed across 15 regions, and the 18 SNPs for maximal strength were distributed across 7 regions.
서 론1990년대 후반부터 발표된 Bouchard et al. [1,2]의 연구에 의하면 스포츠수행력과 관련하여 엘리트 선수의 스포츠수행력의 약 50%가 유전적 요인에 의해 영향을 받는다는 것이며, 최근 대규모 쌍둥이 Genome-wide linkage scan 연구[3]에 의하면 엘리트 선수의 유전적 영향이 약 66%에 해당된다고 제안되었다. 더욱이, Bouchard et al. [1], Fox et al. [4], Lortie et al. [5]에 의하면, 훈련에 대한 개인의 적응능력은 매우 큰 차이를 보이며, 이는 개인의 유전적 요인의 차이에서 비롯된다고 제안되었다. 예를 들면, 유산소성 지구력을 평가하는 주요 지표로 사용되는 최대산소섭취량(VO2max)의 경우 유전적 요인이 약 50%를 차지하며[1,2], 동일한 훈련자극에 대한 개인의 반응 역시 매우 큰 차이를 보인다는 점이다(5-88% 향상됨). 즉, 동일한 훈련자극에 대하여 어떤 사람의 경우는 5%의 매우 낮은 향상을 보이는 반면 어떤 사람은 88%의 급격한 향상을 보인다는 것이며[5], 이러한 개인의 훈련 적응력 또는 향상도의 차이는 유전적 요인의 차이에서 기인하는 것으로 보여진다.
유전학 연구의 핵심 분야는 유전적 표현형(heritable phenotype; 유전자의 작용과 환경에 의해 외부로 나타나는 성질)과 DNA 염기서열변이(sequence variation)의 관련성이다. 즉, 유전적 표현형은 스포츠경기력을 좌우하는 최대산소섭취량(VO2max), 근력, 순발력, 민첩성 등 뿐만 아니라 개인의 체지방률, 신장 등을 의미하며, DNA 염기서열변이는 흔히 말하는 유전자형(genotype)을 의미한다. 따라서 스포츠수행력과 관련이 있는 인간의 DNA 염기서열변이를 찾아낸다면 “운동선수 유전자형(athletic genotype)”을 밝힐 수 있을 것이다.
현재까지 밝혀진 인간의 유전자 수는 약 20,500개로 알려져 있으며[6], 가장 일반적인 DNA 염기서열변이는 단일염기다형성(single nucleotide polymorphism, SNP)으로 대략 300 base pair마다 하나씩 나타난다. 인간의 경우, DNA는 22의 상염색체(autosomes)와 2개의 성염색체 (sex chromosome)에 배열되어 있으며, 각각의 길이는 최소 3천4백만 개 (21번 염색체)에서 최대 2억 6천3백만(1번 염색체) 염기쌍(base pair)으로 총 31억 9천5백5십 개의 염기쌍(base pair, bp)에 이르며[7], 인간이 가지고 있는 총 SNP의 수는 1천만여 개(32억/300 bp)에 이른다[6].
그러나 모든 인간은 염기서열의 99.9%가 똑같은 것으로 나타났으며, 이러한 0.1% SNP(천만여 개×0.1%=10만 개 SNP)의 개인차 때문에 인간은 생물학적으로(질환의 감수성, 외모 등) 조금씩 서로 다르며, 스포츠수행력에 대한 개인차도 마찬가지 원리로 설명될 수 있다.
최근 스포츠수행력 및 건강관련 체력과 관련하여 약 239개의 유전자가 제안되고 있지만[7], 지금까지 제안된 스포츠수행력 및 건강관련 체력과 관련이 있다고 제안된 유전자들을 우리나라의 스포츠영재발굴에 직접적으로 사용하거나 인간의 모든 SNP를 하나씩 분석한다는 것은 엄청난 시간과 경비가 소요되므로 현실적으로 매우 어려운 실정이다. 특히, 스포츠수행력이 단순히 하나의 유전자의 기능으로 결정되기보다는 다유전자의 영향으로 나타나는 표현형으로, 이러한 유전자의 고유기능이 동일 염색체 내(haplotype) 또는 다른 염색체에서 기능이 유사한 다른 유전자형 또는 기능이 상반되는 다른 유전자와의 조합(combination)에 따라 강화되거나 억제될 수 있으며, 이러한 유전자간의 조합이 인종 간(흑인, 백인, 황색인 등) 또는 지역 간(아시아, 유럽, 아프리카 등)에 존재하기 때문이다. 현재 microarray 기술을 이용하여 약 90-100만 개의 SNP를 한 번에 분석할 수 있고[8], 이 기술에 대한 신뢰도와 타당도는 이미 다양한 인종들을 대상으로 한 여러 SNP 연구들에 의해서 입증되었으며, Genome-Wide Human SNP Array를 이용하여 세계적 수준의 우리나라 엘리트 운동선수들(올림픽 참가자/세계선수권대회 메달리스트)의 SNP를 분석한다면 “한국인”만이 가지고 있는 공통적인 유전자형을 밝힐 수 있을 것으로 여겨진다. 따라서 본 연구의 목적은 Genome Wide Association Human SNP Array 6.0을 이용하여 세계적 수준의 우리나라 엘리트 운동선수들의 운동능력별(최대근력 vs. 지구력) SNP를 분석하여, “한국인”만이 가지고 있는 최대근력과 지구력 운동능력별 유전자형을 도출하는 것이며, 이를 통해 “한국형 엘리트선수의 유전자형”을 찾아낼 수 있다면 향후 “체육인재 발굴, 육성”뿐만 아니라 국제 스포츠경쟁력에 크게 이바지할 수 있으리라 기대된다.
연구 방법1. 연구 대상본 연구의 최대근력 집단에 해당하는 역도 종목의 선발대상자는 올림픽 또는 세계선수권 대회에서 메달을 획득한 자로 한정하였으며, 역도 종목의 선발 대상자는 올림픽(남 4명, 여 1명)과 세계선수권 대회(남 10명, 여 3명)에서 메달을 획득한 총 18명 중 본 연구에 자발적으로 참여 의사를 밝힌 16명(35.8±7.0세)이었다. 지구력 집단의 경우, 그동안 마라톤 국제대회에서 2시간 12분 이내의 기록을 보유한 자를 대상으로 한정하였으며, 2시간 12분 이내의 기록을 보유한 한국인은 총 17명으로 이 중 사망, 미국 및 해외 거주자를 제외한 14명 중 자발적으로 본 연구에 참여 의사를 밝힌 남자 12명(41.3±6.9세)이 대상자가 되었다. 그리고 통제집단 또는 비교집단으로 운동선수 경험이 없는 일반 성인 (남자 25명, 여자 1명; 27.8±9.0세)을 대상자로 하였다. 대상자들에게 실험의 목적 및 방법에 대하여 상세히 설명한 후 제반 사항이 포함된 실험참가 서면 동의서 및 IRB 승인(KISS-201308-IF-01)을 받아 연구가 진행되었다.
2. 연구내용본 연구는 심폐지구력을 대표하는 마라톤 종목 최고의 우수 선수들(2시간 12분 이내의 기록 보유자)과 일반인, 그리고 근력(최대근력)을 대표하는 역도 종목 최고의 우수 선수들(올림픽 및 세계선수권 메달리스트)과 일반인들의 genome wide association 연구를 통해 심폐지구력 및 최대근력을 대변할 수 있는 SNPs 도출하였다.
1) DNA 추출DNA 추출은 말초 혈액 샘플을 이용하였으며, 게놈 DNA를 분리한 후 DNA가 추출되었는지를 확인하기 위하여 최종 얻은 투명한 액체(eluent)를 spectrophotometer를 이용, A260/A280 비율에서 DNA의 농도를 확인하였다.
2) Genome-Wide Association Genotyping샘플들은 Affymetrix Genome-Wide Human SNP array 6.0 in the DNALink (Seoul, Korea)를 이용하여 분석하였다. Affymetrix Genome-Wide Human SNP array 6.0은 904,333개 게놈 전체의 SNP와 946,000개의 유전체 단위반복변이(copy number variations)를 포함하였다. 약 500 ng의 게놈 DNA는 Affymetrix의 프로토콜에 따라 두 개의 제한 효소인 NSP I과 Sty I를 이용하여 소화(digested) 처리하였다. 이렇게 소화 처리된 단편들은 보편적인 PCR 프라이밍 순서를 포함하는 특정 어댑터(adaptors)와 결합시켰다. 보편적인 프라이머를 사용한 PCR 증폭은 200-1,100 염기쌍 사이의 단편을 증폭하기 위해 최적화된 반응을 수행하였다. 이미지는 GeneChip의 운영 체제 소프트웨어(Affymetrix, Santa Clara, CA, USA)를 사용하여 분석하였으며, 칩에서 얻은 데이터의 경우, 내부 QC 측정하였다.
DNA 증폭 및 표시. 모든 DNA는 Affymetrix의 Genome-Wide Hu-man SNP Nsp/Sty 6.0 사용 설명서에 따라 genotyping을 준비하였다. 각 DNA의 적정량은 두 개의 중복된 96웰 플레이트로 분할되고, 차례로 각 플레이트의 샘플 48개는 Sty I 또는 NSP 제한 효소로 처리하였다. 각각의 결합된 생성물 중 적정량을 보편적인 primer를 이용하여 PCR 증폭을 위해 네 가지 별도의 플레이트에 나누어 놓았으며, 개별 피험자에 대한 네 개의 PCR 반응의 생성물은 결합 후 플레이트 형식으로 정제하였다. 각각의 증폭된 DNA 샘플의 생성물은 마이크로 플레이트 리더를 이용하여 UV 분광 광도법에 의해 측정하였다. 그 후 각각의 DNA는 DNA 분해효소(partial DNase I digestion)를 이용하여 200 bp보다 작게 세분화 하였고, 세분화된 DNA 조각들의 크기 분포는 아가로스 젤을 이용한 전기영동(4% agarose in Tris-Acetate-EDTA buffer)을 이용하여 평가하였다. 이러한 QC (quality check) 단계를 통과한 샘플들은 Terminal Deoxynucleotidyl Transferase를 이용하여 bioti-nylated nucleoitde와 함께 최종 분류 또는 표시되었으며, 최종 표시된 생성물에 hybridization 용액을 첨가하고, 변성시킨 후 Genome-Wide Human SNP Array 6.0 (1 array/1 sample)에 첨가하였다.
스캔과 genotyping. 배열의 이미지를 획득하기 위하여 각각의 배열을 검사하기 위한 자동 적재기가 포함된 GeneChip® Scanner 3000 7G를 이용하였고, 가공하지 않은(raw) .DAT 파일의 생성을 위하여 GeneChip® Operating System (GCOS) 소프트웨어를 이용하였다. 특징 추출(a feature-extracted, CEL file)을 위하여 각각의 .DAT 이미지는 GCOS를 이용하였다. 모든 .CEL 파일은 Genotyping 사용을 위한 적합성을 결정하기 위하여 Genotyping Console 2.1 (Affymetrix)에서 낮은 수준의 품질 관리(Quality Control, QC) 분석을 실시하였으며, 이 QC 분석은 분명한 제조 또는 물리적 결함이 없음을 확인하기 위한 이미지 품질 검사가 포함되었다. 다음으로, DNA의 품질에 민감한 것으로 알려진 약 3,100의 SNP에 대한 QC Call rate (.CEL 파일이 유전자형 콘솔로 가져올 때 자동으로 생성) 검사를 실시하였다.
Quality Control. 본 연구는 Genome-Wide Human SNP Array 6.0을 이용하여 SNP (Single Nucleotide polymorphism: 단일 핵산염기 다형성) 정보를 확보하였으며, 이러한 대규모 유전체 연구에서는 샘플과 SNP에 대한 적절한 QC가 질적으로 높은 수준의 자료 확보를 결정하므로 중요한 단계라 할 수 있다. 샘플에 대한 QC는 크게 두 단계로 진행되었다. 첫째, 샘플별로 실험 성공률을 뜻하는 call rate이 97% 미만인 샘플은 분석에서 제외하였다. 둘째, 실험을 통해 샘플에 대한 성별 정보가 제시되는데, 실제 임상 정보와 불일치하는 경우 분석에서 제외하였으며, 본 연구에서는 54개 샘플 모두 QC 기준을 통과하여 분석에 포함하였다.
다음으로는 SNP에 대한 QC가 4단계로 진행되었다. 첫째, Hardy-Weinberg Equilibrium (HWE)을 만족하지 않는 SNP는 분석에서 제외하였으며, HWE를 만족하는지 검정하기 위하여 Permutation test를 실시하고, p <.0000001인 경우 분석에서 제외하였다. 둘째, SNP가 각 분석 그룹별 call rate이 95% 미만인 경우는 분석에서 제외하였으며, 셋째, Minor Allele Frequency (MAF)가 각 분석 그룹 모두에서 1% 미만인 경우 분석에서 제외하였다. 마지막으로 통계적 분석을 실시한 후 p-value 가 .0001 미만으로 매우 유의한 결과를 보이는 SNP를 대상으로, genotype 에러를 확인하기 위해 cluster QC plot을 작성하고 QC를 실시하여 genotype 에러인 경우에는 분석에서 제외하였다. 이러한 QC 과정을 통해, 마라톤 vs. 일반인 그리고 역도 선수와 일반인 대상 연구 각각에서 73.85%, 73.09% SNP가 분석에 포함되었다.
3. 자료처리방법SNP가 상염색체(Autosome), 염색체 X 중 Pseudo-autosomal region, 혹은 Mitochondrial에 위치하는 경우, 전체 샘플을 대상으로 또 SNP 가 Y 염색체에 위치하는 경우 남성 대상으로 연관성 연구(Association study)를 실시하였다. 본 연구에서는 여성 샘플이 총 5개로 샘플 사이즈가 충분하지 않아서 X 염색체는 분석에서 제외하였다. SNP는 major 와 minor 두 개의 allele로 구성되며, 각각의 allele을 A와 B라고 한다면 각 샘플이 갖는 genotype은 AA (Major homozygote), AB (Heterozy-gote), BB (Minor homozygote)이다. 이에 대해 다양한 유전적 모형(Genetic model)을 적용하여 통계적 분석을 진행하였다. 본 연구에서는 Dominant (AA vs. AB+BB), Recessive (AA+AB vs. BB), Additive (AA vs. AB vs. BB), 그리고 Allele (A vs. B) 모형을 적용하여 분석하였으며, 모수적 통계방법으로는 Dominant, Recessive, Allele 모형에 대해서는 Chi-square test, Additive 모형에 대해서는 Cochran-Armitage Trend test를 적용하였다. 샘플 사이즈가 충분히 크지 않아서 모수적 방법(Paramet-ric Method)을 적용하기 어려운 경우, 비모수적 방법(Non-Parametric Method)으로 모든 모형에 대해 Jonckheere-Terpstra test를 실시하였다. 모든 통계적 가설 검정은 양측 검정(two-tailed test)을 실시했으며, 통계적 유의수준은 α=0.05로 설정하였다. 통계적 자료분석을 위해서, plink version 1.0.7과 SAS (SAS Institute Inc., Cary, NC) version 9.1.3을 사용하였다.
연구 결과본 연구에 참여한 대상자는 심폐지구력 집단 마라톤 선수 12명(41.3±6.9세), 최대근력집단 역도 선수 16명(35.8±7.0세) 그리고 대조집단 일반인 26명(27.8±9.0세)이었으며, 이들을 대상으로 Genome-Wide Association Study (GWAS)를 통하여 경기력 특성 SNP를 도출하였다.
1. GWAS에 의한 경기력관련 타겟 SNPs 수의 도출1) 분석대상의 총 SNP 개수
Table 1은 본 연구에 참여한 분석대상의 총 SNP 수를 보여주고 있다. 상염색체에 존재하는 분석대상 SNP는 총 867,268개로 전체의 95.9%이었으며, 염색체 X 중 Pseudo-autosomal region에 존재하는 분석대상 SNP는 총 363개로 전체의 0.04%이었고, Mitochondrial에 존재하는 분석대상 SNP는 총 103개로 전체의 0.01%이다. 또한, X 염색체에 존재하는 분석대상 SNP는 총 36,351개로 전체의 4.02%이었고, Y 염색체에 존재하는 분석대상 SNP는 총 248개로 전체의 0.03%이다. X 염색체의 경우, 여성 참여 인원수가 너무 적어 SNPs 집단별 비교, 분석에서는 제외하였다.
2) QC를 통한 최종 분석대상의 SNP 결정최종 분석대상의 SNP 결정을 위한 QC의 결과를 제시하였다. 샘플의 QC에 대한 결과로, 역도선수 16명과 일반인 26명 그리고 마라톤 선수 12명과 일반인 26명을 대상으로 QC 이전의 SNPs는 각각 867,982개에서 QC 후 각각 641,040개(73.85%)와 634,418개(73.09%)가 남았고, 이들 SNPs가 분석되었다.
3) GWAS 결과QC를 통해 결정된 최종 분석대상 SNP 샘플들은 Affymetrix Genome-Wide Human SNP array 6.0 in the DNALink (Seoul, Korea)를 이용하여 Genome-Wide Association을 분석하여 각각의 심페지구력 관련 SNPs와 최대근력 관련 SNPs를 도출하였다. Fig. 1과 2는 마라톤 선수 vs. 일반인, 그리고 역도 선수 vs. 일반인들에 대한 Genome wide association plot (Manhattan Plot)을 제시한 그림이다.
Fig. 1.Fig. 1.Genome wide association plot for marathoners vs. the general population. Horizontal axis: position order by chromosome. Vertical axis: -log10 (p-value) for each SNP. Note that there may be overlap as each dot contains the value of one marker or many markers. Blue line shows p value=.0001. 
Fig. 2.Fig. 2.Genome wide association plot for weightlifters vs. general population. Horizontal axis: position order by chromosome. Vertical axis: -log10 (p-value) for each SNP. Blue line shows p value=.0001. 
Table 2는 p-value 값에 따른 타겟 SNPs의 수를 제시하였다. 본 연구에서는 유의 수준의 역치를 p <.0001을 기준으로 하였으며, 그 결과 최종적으로 심폐지구력 타켓 SNPs (마라톤 선수 vs. 일반인) 총 78개, 최대근력 SNPs (역도 선수 vs. 일반인) 총 45개가 도출되었다.
2. 각 경기력 특성 SNPs의 유전체(Gemonic) 특징1) 심폐지구력 관련 SNPs의 각 구간별 종류 및 위치GWAS를 통하여 선정된 심폐지구력 관련 SNPs는 총 78개였고, p-value<.0001을 만족하는 SNP 중 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간을 한 구간으로 선정하여, 각 구간별 SNPs의 종류 및 위치를 Table 3에 제시하였다.
Table 3.Characteristics of SNPs associated with cardiovascular endurance 심폐지구력 관련 SNPs는 총 78개였으나, 단독으로 존재하는 18개의 SNP 정보는 생략하고, 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간에 있는 60개에 대해서만 요약해서 제시하였다. 또한, 각 SNP의 정확한 위치 및 rs number (the number of human reference SNP clusters)는 본 연구의 중요성과 보안을 위하여 일부 숫자를 부호 처리하여 제시하였다. 심폐지구력 관련 SNPs는 총 15개 구간(100 Kb 거리 이내에 2개 이상의 SNPs를 갖는 구간)에 존재 했으며, 상염색체(Autosome) 1번, 2번, 3번, 7번, 8번, 9번, 10번, 11번, 21번에 존재하는 것으로 나타났다.
2) 최대근력 관련 SNPs의 각 구간별 종류 및 위치GWAS를 통하여 선정된 최대근력 결정 SNPs는 총 45개였고, p-val-ue<.0001을 만족하는 SNPs 중 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간을 선정하여, 각 구간별 SNP의 종류 및 위치를 Table 4에 제시하였다.
Table 4.Characteristics of SNPs associated with maximal strength 최대근력과 관련된 SNPs는 총 45개였으나, 단독으로 존재하는 27개의 SNP 정보는 생략하고, 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간에 있는 18개의 SNPs에 대해서만 요약해서 제시하였다. 각 SNP의 정확한 위치 및 rs number (the number of human reference SNP clusters)는 본 연구의 중요성과 보안을 위하여 일부 숫자를 부호 처리하여 제시하였다. 최대근력과 관련된 SNPs는 총 7개 구간(100 Kb에 2개 이상의 SNPs를 갖는 구간)에 존재 했으며, 상염색체(Autosome) 2번, 7번, 10번, 16번, 18번, 20번에 존재하는 것으로 나타났다.
논 의본 연구는 심폐지구력을 대표하는 마라톤 종목 최고의 우수 선수들(2시간 12분 이내의 기록 보유자)과 일반인, 그리고 근력(최대근력)을 대표하는 역도 종목 최고의 우수 선수들(올림픽 및 세계선수권 메달리스트)과 일반인들의 Genome wide association 연구를 통해 심폐지구력 및 최대근력을 대변할 수 있는 SNPs 도출하였다.
심폐지구력 관련 SNPs는 총 78개였으나, 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간에 있는 60개의 SNPs에 대해서만 Table 3에 제시하였다. 심폐지구력 관련 SNPs는 총 15개 구간(100 Kb 거리 이내에 2개 이상의 SNPs를 갖는 구간)에 존재했으며, 상염색체(Autosome) 1번, 2번, 3번, 7번, 8번, 9번, 10번, 11번, 21번에 존재하는 것으로 나타났다. 이들은 모두 높은 연관비평형(linkage disequilibrium, LD)을 나타내었는데, 이는 이들 SNPs가 염색체 재조합(recombination)이 일어날 때 함께 움직이는(유전되는) 성향을 가지고 있다는 것을 의미한다. 즉, 유전학적인 측면에서 볼 때 심폐지구력이 탁월하게 높은 사람들에게만 특이적으로 이러한 SNPs가 하나의 블록(군집)으로 나타난다는 것으로 해석할 수 있다. 따라서 이러한 SNPs 특히, 심폐지구력의 경우, 60개의 SNPs가 운동 특성을 대변하는 유력한 SNPs일 수 있으며, 이러한 결과는 매우 의미 있는 발견이라 할 수 있다.
최대근력과 관련된 SNPs는 총 45개였으나, 100 Kb 이내에 2개 이상의 SNPs를 갖는 구간에 있는 18개의 SNPs에 대해서만 Table 4에 제시하였다. 최대근력과 관련된 SNPs는 총 7개 구간(100 Kb에 2개 이상의 SNPs를 갖는 구간)에 존재했으며, 상염색체(Autosome) 2번, 7번, 10번, 16번, 18번, 20번에 존재하는 것으로 나타났다. 이들은 모두 높은 연관비평형(LD)을 나타내었는데, 이는 이들 SNPs가 염색체 재조합(recombination)이 일어날 때 함께 움직이는(유전되는) 성향을 가지고 있다는 것을 의미한다. 유전학적인 측면에서 볼 때 최대근력이 우수한 대상자들에게서 특이적으로 이러한 SNPs가 하나의 블록(군집)으로 형성된다는 것으로 해석할 수 있으며, 이러한 최대근력 관련 18개의 SNPs가 최대근력 관련 운동 특성을 대변하는 유력한 SNPs일 수 있는 것이다.
GWAS를 통하여 p-value<.0001을 기준으로 선정된 심폐지구력 관련 SNP는 총 78개였으며, 이들 중 31개가 인트론(intron)에 위치하고 있고, 2개는 엑손(exon)에, 나머지 45개는 상단(upstream) 또는 하단(downstream)에 위치하고 있었다.
인트론의 대부분 서열들은 기능이 없는 DNA라고 여겼지만, 짧은 염기서열의 반복은 효과적인 mRNA절단에 영향을 미친다는 것이 밝혀지면서 전사과정(transcription)에서 스플라이오솜(spliceosome)을 안정화시키며 단백질과 결합하는 부위로 작용하는 것으로 알려져 있다. 한편, 엑손은 최종 단백질을 만들어내는 서열들을 포함하고 있는 부분이며, 유전자의 상단과 하단은 전사과정과 번역과정(translation)을 조절하는 기능을 가지고 있으므로 이 영역에 위치한 SNP는 중요한 생물학적 기능을 가지고 있을 가능성이 매우 높은 것이다. GWAS를 통하여 p-value<.0001을 기준으로 선정된 심폐지구력 관련 78개의 SNP 중 생물학적 기능이 밝혀져 있는 유전자에 위치한 SNP는 55개이다. 본 연구를 통해 밝혀진 심폐지구력 관련 SNPs 중 심폐지구력에 직접적인 영향을 줄 수 있는 생물학적 기능을 가지고 있는 대표적인 SNPs는 다음과 같다.
SNP rsx82042x가 위치한 CACNA1S (calcium channel, voltage-de-pendent, L type, alpha 1S subunit)는 심장근육 조직에서 L-형 변압가변칼슘채널(L-type voltage-dependent calcium channel)을 인코딩(encoding)한다. 세포막 탈분극에 따른 L-형 변압가변칼슘채널의 활성은 심근세포 내 Ca2+의 유입을 일으키고, 이는 근소포체의 Ca2+ 방출을 유발한다. 방출된 Ca2+는 수축성 단백질이 트로포닌 C와 결합하도록 유도하여 심근섬유의 수축이 일어나게 된다[9].
SNP rsx8xxx4x와 rsxxxx4x42가 위치한 CACNA2D1 (calcium channel, voltage-dependent, alpha 2/delta subunit 1)은 L-형 변압가변칼슘채널(L-type voltage-dependent calcium channel)의 알파-2/델타 subunit을 암호화(encoding) 한다. 심장근육세포는 6종류의 아드레날린 수용체를 분출하는데, 3종류의 베타 수용체(베타-1, 베타-2, 베타-3)와 3 종류의 알파 수용체(알파-1A, 알파-1B, 알파-1C)가 있으며, 인간에게는 베타-1 수용체가 지배적인데, 아드레날린에 의한 지속적인 베타-1 수용체에 대한 자극은 G 단백질 결합, cAMP 생성, 그리고 L-형 변압가변칼슘채널의 PKA 인산화에 의해서 심장 수축을 증가시키게 된다[10].
SNP rsx82042x가 위치한 CACNA1S와 SNP rsx8xxx4x와 rsxxxx4x42가 위치한 CACNA2D1은 심장근육조직에서 L-형 변압가변칼슘채널을 인코딩하고, 심근세포 내 Ca2+의 유입을 조절한다. 운동으로 인한 자울신경계의 활성화와 심근섬유의 Ca2+ 민감도 증가는 심근의 수축력을 증가시키고, 결과적으로 좌심실의 비대를 초래하게 된다[9,10].
SNP rsxxxx82x6가 위치한 fms-related tyrosine kinase 4 (FLT4)는 혈관내피세포 성장인자 수용체(vascular endothelial growth factor recep-tor, VEGFR)를 인코딩(encoding) 한다. 혈관내피세포 성장인자(vascular endothelial growth factor) VEGF는 혈관내피세포(endothelial cell)의 세포 분화를 촉진시켜 혈관 형성과 신생을 유발하며, VEGF는 혈관내피세포에 위치한 3개의 수용체(VEGFR-1/Flt-1, VEGFR-2/KDR/Flk-1, VEGFR-3/Flt-4)와 결합하여 활성화된다. 활성화된 VEGFR-3/FLT4는 인산화된 후 STAT3와 STAT5뿐만 아니라 Src, PLC-g, MAPK 등 일련의 세포신호전달 과정을 활성화시켜 혈관 생성을 유발하게 된다[11]. SNP rsxx2xx04가 위치한 MMP2 (matrix metallopeptidase 2)는 (MMP) 단백질 그룹 중 하나이다. MMP는 세포 외 단백질 분해효소(extracel-lular endopeptidases)로서 정상적인 생리적 상태에서 선택적으로 세포 외 기질의 분해에 관여하는데, 기저막의 주요 구조 물질인 type IV 콜라겐을 분해함으로써 혈관생성 조절에 매우 중요한 역할을 담당한다[12].
SNP rs28xxxxx가 위치한 PDE9A (phosphodiesterase 9A)는 phosphodiesterase (PDE) 단백질 그룹 중 하나이다. PDE는 이차전령물질(sec-ond messenger)인 cGMP가 GMP로 가수분해 되는 반응을 촉진시키는 효소로서, 세포 내 신호전달 조절에 매우 중요한 역할을 담당한다. PDE9이 강력한 혈관 이완 인자로 알려진 일산화질소(nitric oxide)의 신호전달 과정에서 cGMP를 GMP로 가수분해 시킴으로써 cGMP의 활성을 감소시켜 혈관 이완을 조절하는 기전을 나타낸다. PDE가 심장세포에서 cGMP의 활성을 저해함으로써 Protein Kinase G (PKG)를 감소시켜 결과적으로 심근의 수축을 감소되는 기전을 보이게 된다[13].
SNP rsx68x842x가 위치한 FBXO32 (F-box protein 32)는 forkhead box O (FOXO) 단백질 그룹 중 하나이다. FOXO 단백질은 전사인자 (transcription factor)로서 세포 자멸(apoptosis), 세포 주기 조절 (cell-cy-cle control), 당 대사(glucose metabolism), 산화스트레스 저항(oxidative stress resistance), 근 위축(muscle atrophy) 등에 관여하는 유전자들의 발현을 조절한다. FOXO 단백질은 serine-threonine kinase Akt/protein kinase B (Akt/PKB)와 phosphatidylinositol 3-kinase (PI3K)의 인산화에 의해서 조절된다[14].
GWAS를 통하여 p-value<.0001을 기준으로 선정된 최대근력 관련 45개의 SNP 중 생물학적 기능이 밝혀져 있는 유전자에 위치한 SNP은 41개이며, 이들 중 23개가 인트론(intron)에 위치하고 있고, 3개는 엑손(exon)에, 나머지 19개는 상단(upstream) 또는 하단(downstream)에 위치하고 있었다.
앞서 언급한 바와 같이 인트론 역시 mRNA절단뿐만 아니라 전사과정(transcription)에서의 스플라이오솜(spliceosome)의 안정화 그리고 단백질과 결합하는 부위로 작용하는 것으로 알려져 있고, 엑손은 최종 단백질을 만들어내는 서열들을 포함하고 있는 부분이며, 유전자의 상단과 하단은 전사과정과 번역과정(translation)을 조절하는 기능을 가지고 있으므로 이 영역에 위치한 SNP는 중요한 생물학적 기능을 가지고 있을 가능성이 매우 높은 것이다. 본 연구를 통해 밝혀진 최대근력 관련 SNPs 중 최대근력에 직접적인 영향을 줄 수 있는 생물학적 기능을 가지고 있는 대표적인 SNPs는 다음과 같다.
세포의 성장 및 분화, 대사작용, 생식, 노화와 같은 기본적인 생리현상들은 세포들 간의 유기적인 정보 교환에 의해 조절되고 유지되며, 이는 외부인자(신경전달물질, 호르몬, 성장인자 등)에 의한 신호가 세포막에 존재하는 특이적인 수용체에 의해 세포 내로 전달되고 증폭됨으로써 이루어진다. 따라서, 세포들 간의 유기적인 신호전달은 근육 생성, 신경계 발달, 혈관 생성, 신진대사 조절 등에 관여하므로 최대근력에 직접적인 영향을 줄 수 있는 것이다. 신호전달 수용체는 채널과 연계된 수용체(이온채널 수용체), 효소활성과 연계된 수용체(예: 단백질 키나아제), G-단백질과 연계된 수용체와 같이 세포막에 존재하는 수용체가 있고, 세포막을 통과하기 어려운 물질을 운반하는 운반체(예: 단백질 운반체)가 있다. 수용체에 의해 세포 내로 전달된 신호는 주로 이차전령(예: cGMP, cAMP), 신호전달 단백질, 조절 단백질 간의 일련의 화학 반응(예: 인산화)을 통해서 목표 물질(예: 핵, DNA)까지 전달되는 것이다.
최대근력 관련 SNP 중 15개 SNP (rsx868x0x, rs26x0xx8, rs4x8xxxx, rsxxxx82x6, rs42x66xx, rsxxx6x6xx, rsxxxx04xx, rsx6x24xx6, rs2240xx, rsx08x8x6x, rsxxxxxxx0, rsx08868x2, rsxx6x64x, rsx826644, rs6xxxxx4)가 신호전달 과정과 관련이 있는 유전자(THRB, LPHN3, TMEM167A, FLT4, SLC13A4, DPP6, DMRT1, SLC24A2, GPR144, PSMB7, FGFR2, ABAT, DLGAP1, SNX5)에 위치하고 있다. SNP rsx868x0x가 위치한 THRB는 갑상선 호르몬(thyroid hormone)의 신호전달경로에 관여할 뿐만 아니라 세포 간 소통, 세포 주기 조절, 성장 조절, 근 수축, 탄수화물 및 지질의 신진대사 등 다양한 생물학적 기능에 관여하게 된다[15].
근육 세포(myocyte)는 근섬유로 이루어져 있으며, 이는 트로포미오신, 트로포닌 등과 같은 단백질을 함유하고 있는 액틴과 미오신으로 이루어져 있다. 섬유아세포(fibroblast)는 섬유 모세포 등으로 이루어진 증식력이 강한 어린 결합 조직으로 세포 외 기질과 콜라겐을 생성하여 근육 조직의 구조적 골조를 형성한다. 신경근접합부(neuromuscular junction)는 운동신경종말과 근섬유를 연결하는 부위로 축삭세포막과 근세포막은 시냅스를 이루고 있으며, 아세틸콜린과 같은 자극전달물질에 의해 전기적 자극이 시냅스전막을 통해 근세포막에 전달되면 근섬유는 수축하게 되고 이때 근력이 발생하게 된다. 최대근력 관련 SNP 중 18개 SNP (rs2x628x6, rs6xx2x0x, rsxx04xx00, rsx0x8xx2x, rsxx04xx06, rsx04x4x2x, rsx02x4xxx, rsx826xx8, rsx006xxx, rsx0x0xxx0, rsx0x4044x, rsxx0x0xx, rsxxxxxxx0, rsx08868x2, rsxxx8xxx, rsx0x6xxxx, rsxxx82xx, rs6x06xxx)는 근육 조직의 형성 및 발달 과정과 관련이 있는 유전자 (PADI6, MYCN, HS6ST1, COBL, KCNU1, ZNF703, VLDLR, SMAR-CA2, ADK, FGFR2, CDH11, PTPRM)에 위치하고 있다. SNP rsxxx8xxx 와 rsx0x6xxxx가 위치한 CDH11은 액틴 Rho family 신호전달 경로 재생, 근세포 형성, 세포 분화 등을 조절하는 Rho 그룹의 신호전달에 관여한다[16].
신경 세포(neuron)는 핵이 있는 신경세포체와 다른 세포에서 신호를 받는 수상돌기, 그리고 다른 세포에 신호를 주는 축삭돌기로 이루어져 있고 돌기 사이에 신호를 전달하는 부분이 시냅스이며 그 역할에 따라 감각뉴런, 연합뉴런, 운동뉴런의 세 종류가 있다. 운동뉴런은 근섬유와 시냅스를 이루어 신경근접합부(neuromuscular junction)를 형성함으로써 근 수축을 유발하게 된다. 최대근력 관련 SNP 중 18개 SNP (rsxx04xx00, rsx0x8xx2x, rsxx04xx06, rsx04x4x2x, rsx868x0x, rs26x0xx8, rsx88xx2x, rs4xx4x26, rsx08x8x6x, rsxxxxxxx0, rsx08868x2, rsxx2xx6x, rs6x00xxx, rsxx6x64x, rsx826644, rs60xx8xx, rs60xx844, rs60xxxxx)가 신경 조직의 형성 및 발달 과정과 관련이 있는 유전자 (HS6ST1, THRB, LPHN3, CNTNAP2, NTRK2, GPR144, PSMB7, FGFR2, COL4A2, RBFOX1, ABAT, DLGAP1, ATRN)에 위치하고 있다. SNP rs4xx4x26가 위치한 NTRK2는 tropomyosin-related kinase (TRK) family 중 하나로 nerve growth factor (NGF), brain-derived neurotroph-ic factor (BDNF), neurotrophin-3 (NT-3), neurotrophin-4 (NT-4) 등과 결합하여 Ras/Raf/MEK/ERK 경로와 PI3K/AKT 경로를 활성화시키는 신호를 전달함으로써 신경세포의 분화를 촉진하고 중추와 말초신경계의 신경세포 기능을 향상시키게 된다[17].
생체의 모든 세포는 생명활동을 위해 당질, 지질, 단백질을 외부로부터 섭취하여 내부에 축적하고 생체 내에 화학반응에 이용할 수 있는 형태로 동화 또는 이화하는 과정을 필요로 한다. 특히, 근 수축 시 최대 근력을 유발하기 위해서는 이러한 일련의 과정이 매우 유기적이고 효과적으로 일어나야한다. 최대근력 관련 SNP 중 15개 SNP (rsxx04xx00, rsx0x8xx2x, rsxx04xx06, rsx04x4x2x, rsx2xx4x4x, rsx868x0x, rsx0xxx4, rsx006xxx, SMARCA2, rsx08x8x6x, rsx0x0xxx0, rsx0x4044x, rsxx0x0xx, rsxxx82xx, rs6x06xxx)는 신진대사에 관련된 유전자(HS6ST1, VENTXP7, THRB, DENND3, VLDLR, SMARCA2, GPR144, PSMB7, ADK, PTPRM)에 위치하고 있다. SNP rsxx04xx00, rsx0x8xx2x, rsxx04xx06, rsx04x4x2x 가 위치한 HS6ST1이 탄수화물 신진대사와 관련해서 여러 유전자, 단백질, 효소들과 복잡하게 연결되어있다. 이러한 네트워크 현상으로 섭취한 당질로부터 생합성을 위해 에너지와 탄소를 추출해내는 과정, 체내에 글리코겐 형태로 당을 잠시 저장하는 과정, 그리고 좀 더 긴 시간 동안 피루브산으로부터 당을 합성하는 과정을 가능하게 해주는 것이다[18].
최대근력 관련 SNP 중 8개 SNP (rsxx04xx00, rsx0x8xx2x, rsxx04xx06, rsx04x4x2x, rsxxxx82x6, rsx826xx8, rsxxxxxxx0, rsx08868x2)는 혈관 생성, 사지(팔, 다리) 형성 및 성장에 관련된 유전자(HS6ST1, FLT4, KCNU1, ZNF70, FGFR2)에 위치하고 있다. SNP rsxxxx82x6가 위치한 FLT4는 혈관내피성장인자(VEGF)의 수용체 역할을 담당하며, 인체 내에서 혈관생성을 위한 주요한 역할을 담당하고 있으며, FLT4 신호전달은 CASP1 (caspase 1) 의존성 인플라마좀 활성화를 억제하면서도 박테리아 제거를 위한 MAP1LC3/LC3 활성화를 높여주는 역할을 담당한다[19].
결 론결론적으로 본 연구를 통해 Genome-Wide Human SNP Array 6.0을 이용하여 밝혀진 심폐지구력 관련 60개의 SNPs과 최대근력 관련 18개의 SNPs가 운동 특성을 대변하는 유력한 SNPs일 수 있으며, 향후 “한국인”만이 가지고 있는 특이적인 유전자형을 도출해가는데 중요한 정보를 제공하였다고 사료된다. 본 연구의 취약점이자 강점은 종목 특성별 국내 최우수 선수들만을 대상하였다는 점이다. 즉, 올림픽과 세계선수권에서, 메달을 획득한 최우수 선수들만을 대상으로 하였기에 경기력 특성을 가장 잘 반영할 수 있는 동질성 확보라는 측면에서 장점이 있으나 샘플 수가 매우 제한적이라는 한계를 지니고 있다는 점 그리고 비교집단의 샘플 수가 적다는 점이다. 추후 연구에서는 이들 집단의 샘플 수를 확대하여 추가 검증할 필요가 있다.
REFERENCES1. Bouchard C, Daw EW, Rice T, Perusse L, Gagnon J, et al. Familial resemblance for VO2max in the sedentary state: the HERITAGEfamily study. Med Sci Sports Exerc. 1998;30(2):252-8.
2. Bouchard C, Rankinen T, Chagnon YC, Rice T, Perusse L, et al. Genomic scan for maximal oxygen uptake and its response to training inthe HERITAGE family study. J Appl Physiol. 2000;88(2):551-9.
3. De Moor MH, Posthuma D, Hottenga JJ, Willemsen G, Boomsma DI, et al. Genome-wide linkage scan for exercise participation in dutch sibling pairs. Eur J Hum Genet. 2007;15(12):1252-9.
4. Fox PW, Hershberger SL, Bouchard TJ Jr. Genetic and environmental contributions to the acquisition of a motor skill. Nature. 1996;384(6607):356-8.
5. Lortie G, Simoneau JA, Hamel P, Boulay MR, Landry F, et al. Re-sponses of maximal aerobic power and capacity to aerobic training. Int J Sports Med. 1984;5(5):232-6.
7. Bray MS, Hagberg JM, Perusse L, Rankinen T, Roth SM, et al. The human gene map for performance and health-related fitnessphenotypes: the 2006-2007 update. Med Sci Sports Exerc. 2009;41(1):35-73.
8. Lippi G, Longo UG, Maffulli N. Genetics and sports. Br Med Bull (Epub ahead of print). 2009;1-21.
9. Teresa AB, Emily FL, David FK. Pharmacogenetics and pathophysiol-ogy of CACNA1S mutations in malignant hyperthermia. Physiol Genomics. 2017;149(2):81-7.
10. Dahimene S, Elsner L, Holling T, Mattas LS, Pickard J, et al. Biallelic CACNA2D1 loss-of-function variants cause early-onset developmental epileptic encephalopathy. Case Reports Brain. 2022;145(8):2721-9.
11. Ma L, Li W, Zhang Y, Qi L, Zhao Q, et al. FLT4/VEGFR3 activates AMPK to coordinate glycometabolic reprogramming with autophagy and inflammasome activation for bacterial elimination. Autophagy. 2022;18(6):1385-400.
12. Liu D, Kang H, Gao M, Jin L, Zhang F, et al. Exosome-transmitted circ_MMP2 promotes hepatocellular carcinoma metastasis by upregu-lating MMP2. Mol Oncol. 2020;14(6):1365-80.
13. Kolb TM, Johnston L, Damarla M, Kass DA, Hassoun PM. PDE9Ad-eficiency does not prevent chronic-hypoxic pulmonary hypertension in mice. Physiol Rep. 2021;9(18):e15057.
14. Mei Z, Zhang D, Hu B, Wang J, Shen X, et al. FBXO32 targets c-myc for proteasomal degradation and inhibits c-myc activity. Biol Chem. 2015;290(26):16202-14.
15. Zhou Z, Yang C, Fuyan Lv, Liu W, Yan S, et al. Novel THRB mutation analysis in congenital hypothyroidism with thyroid dysgenesis. J Cell Biochem. 2018;119(11):9474-82.
16. Ruan W, Pan R, Shen X, Nie Y, Wu Y. CDH11 promotes liver fibrosis via activation of hepatic stellate cells. Biochem Biophys Res Commun. 2019;508(2):543-9.
17. Lee HC, Lin SC, Wu MH, Tsa SJ. Inhibiting NTRK2 signaling causes endometriotic lesion regression. Reproduction. 2021;161(1):11-9.
|
|
|||||||||||||||||||||||||||||||||||||||